Nathalialisboarosaalmeidago
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

Accepted Version
Article Genetic Control of Gonadal Sex Determination and Development STEVANT, Isabelle, NEF, Serge Abstract Sex determination is the process by which the bipotential gonads develop as either testes or ovaries. With two distinct potential outcomes, the gonadal primordium offers a unique model for the study of cell fate specification and how distinct cell populations diverge from multipotent progenitors. This review focuses on recent advances in our understanding of the genetic programs and epigenetic mechanisms that regulate gonadal sex determination and the regulation of cell fate commitment in the bipotential gonads. We rely primarily on mouse data to illuminate the complex and dynamic genetic programs controlling cell fate decision and sex-specific cell differentiation during gonadal formation and gonadal sex determination. Reference STEVANT, Isabelle, NEF, Serge. Genetic Control of Gonadal Sex Determination and Development. Trends in Genetics, 2019 PMID : 30902461 DOI : 10.1016/j.tig.2019.02.004 Available at: http://archive-ouverte.unige.ch/unige:115790 Disclaimer: layout of this document may differ from the published version. 1 / 1 Trends in Genetics Genetic control of sex determination and gonad development --Manuscript Draft-- Manuscript Number: TIGS-D-18-00173R1 Article Type: Review Keywords: sex determination; ovary; testis; lineage specification; gene expression; epigenetic regulation Corresponding Author: Serge Nef geneva, SWITZERLAND First Author: Isabelle Stévant Order of Authors: Isabelle Stévant Serge Nef Abstract: Sex determination is the process by which the bipotential gonads develop as either testes or ovaries. With two distinct potential outomes, the gonadal primordium offers a unique model for the study of cell fate specification and how distinct cell populations diverge from multipotent progenitors. -

Analysis of the Indacaterol-Regulated Transcriptome in Human Airway
Supplemental material to this article can be found at: http://jpet.aspetjournals.org/content/suppl/2018/04/13/jpet.118.249292.DC1 1521-0103/366/1/220–236$35.00 https://doi.org/10.1124/jpet.118.249292 THE JOURNAL OF PHARMACOLOGY AND EXPERIMENTAL THERAPEUTICS J Pharmacol Exp Ther 366:220–236, July 2018 Copyright ª 2018 by The American Society for Pharmacology and Experimental Therapeutics Analysis of the Indacaterol-Regulated Transcriptome in Human Airway Epithelial Cells Implicates Gene Expression Changes in the s Adverse and Therapeutic Effects of b2-Adrenoceptor Agonists Dong Yan, Omar Hamed, Taruna Joshi,1 Mahmoud M. Mostafa, Kyla C. Jamieson, Radhika Joshi, Robert Newton, and Mark A. Giembycz Departments of Physiology and Pharmacology (D.Y., O.H., T.J., K.C.J., R.J., M.A.G.) and Cell Biology and Anatomy (M.M.M., R.N.), Snyder Institute for Chronic Diseases, Cumming School of Medicine, University of Calgary, Calgary, Alberta, Canada Received March 22, 2018; accepted April 11, 2018 Downloaded from ABSTRACT The contribution of gene expression changes to the adverse and activity, and positive regulation of neutrophil chemotaxis. The therapeutic effects of b2-adrenoceptor agonists in asthma was general enriched GO term extracellular space was also associ- investigated using human airway epithelial cells as a therapeu- ated with indacaterol-induced genes, and many of those, in- tically relevant target. Operational model-fitting established that cluding CRISPLD2, DMBT1, GAS1, and SOCS3, have putative jpet.aspetjournals.org the long-acting b2-adrenoceptor agonists (LABA) indacaterol, anti-inflammatory, antibacterial, and/or antiviral activity. Numer- salmeterol, formoterol, and picumeterol were full agonists on ous indacaterol-regulated genes were also induced or repressed BEAS-2B cells transfected with a cAMP-response element in BEAS-2B cells and human primary bronchial epithelial cells by reporter but differed in efficacy (indacaterol $ formoterol . -

Investigation of KRAS Dependency Bypass and Functional Characterization of All Possible KRAS Missense Variants
Investigation of KRAS Dependency Bypass and Functional Characterization of All Possible KRAS Missense Variants The Harvard community has made this article openly available. Please share how this access benefits you. Your story matters Citable link http://nrs.harvard.edu/urn-3:HUL.InstRepos:40050098 Terms of Use This article was downloaded from Harvard University’s DASH repository, and is made available under the terms and conditions applicable to Other Posted Material, as set forth at http:// nrs.harvard.edu/urn-3:HUL.InstRepos:dash.current.terms-of- use#LAA Investigation of KRAS Dependency Bypass and Functional Characterization of All Possible KRAS Missense Variants A dissertation presented by Seav Huong Ly to The Division of Medical Sciences in partial fulfillment of the requirements for the degree of Doctor of Philosophy in the subject of Biological and Biomedical Sciences Harvard University Cambridge, Massachusetts April 2018 © 2018 Seav Huong Ly All rights reserved. Dissertation Advisor: William C. Hahn Seav Huong Ly Investigation of KRAS Dependency Bypass and Functional Characterization of All Possible KRAS Missense Variants Abstract The importance of oncogenic KRAS in human cancers have prompted intense efforts to target KRAS and its effectors. To anticipate the development of resistance to these strategies, we previously performed a genome-scale expression screen to identify genes that bypass KRAS oncogenic dependency. Here we test thirty-seven genes that scored over five standard deviations and find that overexpression of LIM homeobox 9 (LHX9), a transcription factor involved in embryonic development, robustly rescues the suppression of KRAS in vitro and xenograft models. Furthermore, LHX9 substantially decreases cell sensitivity to KRASG12C and MEK1/2 inhibitors in KRAS-dependent cells. -
76F0c47cb71e753c0f29618f48ed
International Journal of Molecular Sciences Review Strategies and Challenges to Improve Cellular Programming-Based Approaches for Heart Regeneration Therapy Lin Jiang, Jialiang Liang , Wei Huang, Zhichao Wu, Christian Paul and Yigang Wang * Department of Pathology and Laboratory Medicine, College of Medicine, University of Cincinnati Medical Center, Cincinnati, OH 45267-0529, USA; [email protected] (L.J.); [email protected] (J.L.); [email protected] (W.H.); [email protected] (Z.W.); [email protected] (C.P.) * Correspondence: [email protected]; Tel.: +1-513-558-5798 Received: 30 September 2020; Accepted: 15 October 2020; Published: 16 October 2020 Abstract: Limited adult cardiac cell proliferation after cardiovascular disease, such as heart failure, hampers regeneration, resulting in a major loss of cardiomyocytes (CMs) at the site of injury. Recent studies in cellular reprogramming approaches have provided the opportunity to improve upon previous techniques used to regenerate damaged heart. Using these approaches, new CMs can be regenerated from differentiation of iPSCs (similar to embryonic stem cells), the direct reprogramming of fibroblasts [induced cardiomyocytes (iCMs)], or induced cardiac progenitors. Although these CMs have been shown to functionally repair infarcted heart, advancements in technology are still in the early stages of development in research laboratories. In this review, reprogramming-based approaches for generating CMs are briefly introduced and reviewed, and the challenges (including low efficiency, functional maturity, and safety issues) that hinder further translation of these approaches into a clinical setting are discussed. The creative and combined optimal methods to address these challenges are also summarized, with optimism that further investigation into tissue engineering, cardiac development signaling, and epigenetic mechanisms will help to establish methods that improve cell-reprogramming approaches for heart regeneration. -
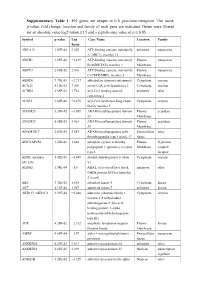
Supplementary Table 1
Supplementary Table 1. 492 genes are unique to 0 h post-heat timepoint. The name, p-value, fold change, location and family of each gene are indicated. Genes were filtered for an absolute value log2 ration 1.5 and a significance value of p ≤ 0.05. Symbol p-value Log Gene Name Location Family Ratio ABCA13 1.87E-02 3.292 ATP-binding cassette, sub-family unknown transporter A (ABC1), member 13 ABCB1 1.93E-02 −1.819 ATP-binding cassette, sub-family Plasma transporter B (MDR/TAP), member 1 Membrane ABCC3 2.83E-02 2.016 ATP-binding cassette, sub-family Plasma transporter C (CFTR/MRP), member 3 Membrane ABHD6 7.79E-03 −2.717 abhydrolase domain containing 6 Cytoplasm enzyme ACAT1 4.10E-02 3.009 acetyl-CoA acetyltransferase 1 Cytoplasm enzyme ACBD4 2.66E-03 1.722 acyl-CoA binding domain unknown other containing 4 ACSL5 1.86E-02 −2.876 acyl-CoA synthetase long-chain Cytoplasm enzyme family member 5 ADAM23 3.33E-02 −3.008 ADAM metallopeptidase domain Plasma peptidase 23 Membrane ADAM29 5.58E-03 3.463 ADAM metallopeptidase domain Plasma peptidase 29 Membrane ADAMTS17 2.67E-04 3.051 ADAM metallopeptidase with Extracellular other thrombospondin type 1 motif, 17 Space ADCYAP1R1 1.20E-02 1.848 adenylate cyclase activating Plasma G-protein polypeptide 1 (pituitary) receptor Membrane coupled type I receptor ADH6 (includes 4.02E-02 −1.845 alcohol dehydrogenase 6 (class Cytoplasm enzyme EG:130) V) AHSA2 1.54E-04 −1.6 AHA1, activator of heat shock unknown other 90kDa protein ATPase homolog 2 (yeast) AK5 3.32E-02 1.658 adenylate kinase 5 Cytoplasm kinase AK7 -

Down-Regulation Networks in Acute Strenuous Exercise
Transcriptional profile in rat muscle: down-regulation networks in acute strenuous exercise Stela Mirla da Silva Felipe1,*, Raquel Martins de Freitas1, Emanuel Diego dos Santos Penha1, Christina Pacheco1, Danilo Lopes Martins2, Juliana Osório Alves1, Paula Matias Soares1, Adriano César Carneiro Loureiro1, Tanes Lima3, Leonardo R. Silveira3, Alex Soares Marreiros Ferraz1, Jorge Estefano Santana de Souza2, Jose Henrique Leal-Cardoso1, Denise P. Carvalho4 and Vania Marilande Ceccatto1,* 1 Superior Institute of Biomedic Sciences, Universidade Estadual do Ceará, Fortaleza, Ceará, Brazil 2 Digital Metropolis Institute, Universidade Federal do Rio Grande do Norte, Natal, Rio Grande do Norte, Brazil 3 Institute of Biology, Universidade Estadual de Campinas, Campinas, São Paulo, Brazil 4 Carlos Chagas Filho Biophysics Institute, Universidade Federal do Rio de Janeiro, Rio de Janeiro, Brazil * These authors contributed equally to this work. ABSTRACT Background. Physical exercise is a health promotion factor regulating gene expression and causing changes in phenotype, varying according to exercise type and intensity. Acute strenuous exercise in sedentary individuals appears to induce different transcrip- tional networks in response to stress caused by exercise. The objective of this research was to investigate the transcriptional profile of strenuous experimental exercise. Methodology. RNA-Seq was performed with Rattus norvegicus soleus muscle, submit- ted to strenuous physical exercise on a treadmill with an initial velocity of 0.5 km/h and increments of 0.2 km/h at every 3 min until animal exhaustion. Twenty four hours post-physical exercise, RNA-seq protocols were performed with coverage of 30 million reads per sample, 100 pb read length, paired-end, with a list of counts totaling 12816 Submitted 26 May 2020 genes. -

KIF14 Is a Candidate Oncogene in the 1Q Minimal Region of Genomic Gain in Multiple Cancers
Oncogene (2005) 24, 4741–4753 & 2005 Nature Publishing Group All rights reserved 0950-9232/05 $30.00 www.nature.com/onc ORIGINAL PAPERS KIF14 is a candidate oncogene in the 1q minimal region of genomic gain in multiple cancers Timothy W Corson1,2, Annie Huang3, Ming-Sound Tsao4,5,6 and Brenda L Gallie*,1,2,5,7 1Division of Cancer Informatics, Ontario Cancer Institute/Princess Margaret Hospital, University Health Network, Toronto, ON, Canada M5G 2M9; 2Department of Molecular & Medical Genetics, University of Toronto, Toronto, ON, Canada M5S 1A8; 3Labatt Brain Tumour Research Centre, Cancer Research Program, Hospital for Sick Children, Toronto, ON, Canada M5G 1X8; 4Division of Cellular & Molecular Biology, Ontario Cancer Institute/Princess Margaret Hospital, University Health Network, Toronto, ON, Canada M5G 2M9; 5Department of Medical Biophysics, University of Toronto, Toronto, ON, Canada M5G 2M9; 6Department of Laboratory Medicine and Pathobiology, University of Toronto, Toronto, ON, Canada M5G 1L5; 7Department of Ophthalmology, University of Toronto, Toronto, ON, Canada M5G 1X5 Gain of chromosome 1q31–1q32is seen in >50% of in retinoblastoma development is the loss of function of retinoblastoma and is common in other tumors. To define both alleles of the prototypic tumor suppressor gene, the minimal 1q region of gain, we determined genomic RB1, which encodes a key cell-cycle negative regulatory copy number by quantitative multiplex PCR of 14 transcription factor, pRB (Classon and Harlow, 2002). sequence tagged sites (STSs) spanning 1q25.3–1q41. The These initiating ‘M1’ and ‘M2’ mutations of Knudson’s most frequently gained STS at 1q32.1 (71%; 39 of 55 classic two-hit model for oncogenesis (Knudson, 1971) retinoblastoma) defined a 3.06 Mbp minimal region of are necessary for retinoblastoma initiation, but not gain between flanking markers, containing 14 genes. -

Novel IQCE Variations Confirm Its Role in Postaxial Polydactyly and Cause
Novel IQCE variations confirm its role in postaxial polydactyly and cause ciliary defect phenotype in zebrafish Alejandro Estrada-Cuzcano, Christelle Etard, Clarisse Delvallée, Corinne Stoetzel, Elise Schaefer, Sophie Scheidecker, Véronique Geoffroy, Aline Schneider, Fouzia Studer, Francesca Mattioli, et al. To cite this version: Alejandro Estrada-Cuzcano, Christelle Etard, Clarisse Delvallée, Corinne Stoetzel, Elise Schaefer, et al.. Novel IQCE variations confirm its role in postaxial polydactyly and cause ciliary defect phenotype in zebrafish. Human Mutation, Wiley, 2019, 10.1002/humu.23924. hal-02304111 HAL Id: hal-02304111 https://hal.archives-ouvertes.fr/hal-02304111 Submitted on 2 Oct 2019 HAL is a multi-disciplinary open access L’archive ouverte pluridisciplinaire HAL, est archive for the deposit and dissemination of sci- destinée au dépôt et à la diffusion de documents entific research documents, whether they are pub- scientifiques de niveau recherche, publiés ou non, lished or not. The documents may come from émanant des établissements d’enseignement et de teaching and research institutions in France or recherche français ou étrangers, des laboratoires abroad, or from public or private research centers. publics ou privés. Page 1 of 125 Human Mutation Novel IQCE variations confirm its role in postaxial polydactyly and cause ciliary defect phenotype in zebrafish. Alejandro Estrada-Cuzcano1*, Christelle Etard2*, Clarisse Delvallée1*, Corinne Stoetzel1, Elise Schaefer1,3, Sophie Scheidecker1,4, Véronique Geoffroy1, Aline Schneider1, Fouzia Studer5, Francesca Mattioli6, Kirsley Chennen1,7, Sabine Sigaudy8, Damien Plassard9, Olivier Poch7, Amélie Piton4,6, Uwe Strahle2, Jean Muller1,4* and Hélène Dollfus1,3,5* 1. Laboratoire de Génétique médicale, UMR_S INSERM U1112, IGMA, Faculté de Médecine FMTS, Université de Strasbourg, Strasbourg, France. -

BMC Biology Biomed Central
BMC Biology BioMed Central Research article Open Access Classification and nomenclature of all human homeobox genes PeterWHHolland*†1, H Anne F Booth†1 and Elspeth A Bruford2 Address: 1Department of Zoology, University of Oxford, South Parks Road, Oxford, OX1 3PS, UK and 2HUGO Gene Nomenclature Committee, European Bioinformatics Institute (EMBL-EBI), Wellcome Trust Genome Campus, Hinxton, Cambridgeshire, CB10 1SA, UK Email: Peter WH Holland* - [email protected]; H Anne F Booth - [email protected]; Elspeth A Bruford - [email protected] * Corresponding author †Equal contributors Published: 26 October 2007 Received: 30 March 2007 Accepted: 26 October 2007 BMC Biology 2007, 5:47 doi:10.1186/1741-7007-5-47 This article is available from: http://www.biomedcentral.com/1741-7007/5/47 © 2007 Holland et al; licensee BioMed Central Ltd. This is an Open Access article distributed under the terms of the Creative Commons Attribution License (http://creativecommons.org/licenses/by/2.0), which permits unrestricted use, distribution, and reproduction in any medium, provided the original work is properly cited. Abstract Background: The homeobox genes are a large and diverse group of genes, many of which play important roles in the embryonic development of animals. Increasingly, homeobox genes are being compared between genomes in an attempt to understand the evolution of animal development. Despite their importance, the full diversity of human homeobox genes has not previously been described. Results: We have identified all homeobox genes and pseudogenes in the euchromatic regions of the human genome, finding many unannotated, incorrectly annotated, unnamed, misnamed or misclassified genes and pseudogenes. -

(12) Patent Application Publication (10) Pub. No.: US 2009/0269772 A1 Califano Et Al
US 20090269772A1 (19) United States (12) Patent Application Publication (10) Pub. No.: US 2009/0269772 A1 Califano et al. (43) Pub. Date: Oct. 29, 2009 (54) SYSTEMS AND METHODS FOR Publication Classification IDENTIFYING COMBINATIONS OF (51) Int. Cl. COMPOUNDS OF THERAPEUTIC INTEREST CI2O I/68 (2006.01) CI2O 1/02 (2006.01) (76) Inventors: Andrea Califano, New York, NY G06N 5/02 (2006.01) (US); Riccardo Dalla-Favera, New (52) U.S. Cl. ........... 435/6: 435/29: 706/54; 707/E17.014 York, NY (US); Owen A. (57) ABSTRACT O'Connor, New York, NY (US) Systems, methods, and apparatus for searching for a combi nation of compounds of therapeutic interest are provided. Correspondence Address: Cell-based assays are performed, each cell-based assay JONES DAY exposing a different sample of cells to a different compound 222 EAST 41ST ST in a plurality of compounds. From the cell-based assays, a NEW YORK, NY 10017 (US) Subset of the tested compounds is selected. For each respec tive compound in the Subset, a molecular abundance profile from cells exposed to the respective compound is measured. (21) Appl. No.: 12/432,579 Targets of transcription factors and post-translational modu lators of transcription factor activity are inferred from the (22) Filed: Apr. 29, 2009 molecular abundance profile data using information theoretic measures. This data is used to construct an interaction net Related U.S. Application Data work. Variances in edges in the interaction network are used to determine the drug activity profile of compounds in the (60) Provisional application No. 61/048.875, filed on Apr. -

Program Book
The Genetics Society of America Conferences 15th International Xenopus Conference August 24-28, 2014 • Pacific Grove, CA PROGRAM GUIDE LEGEND Information/Guest Check-In Disabled Parking E EV Charging Station V Beverage Vending Machine N S Ice Machine Julia Morgan Historic Building W Roadway Pedestrian Pathway Outdoor Group Activity Area Program and Abstracts Meeting Organizers Carole LaBonne, Northwestern University John Wallingford, University of Texas at Austin Organizing Committee: Julie Baker, Stanford Univ Chris Field, Harvard Medical School Carmen Domingo, San Francisco State Univ Anna Philpott, Univ of Cambridge 9650 Rockville Pike, Bethesda, Maryland 20814-3998 Telephone: (301) 634-7300 • Fax: (301) 634-7079 E-mail: [email protected] • Web site: genetics-gsa.org Thank You to the Following Companies for their Generous Support Platinum Sponsor Gold Sponsors Additional Support Provided by: Carl Zeiss Microscopy, LLC Monterey Convention & Gene Tools, LLC Visitors Bureau Leica Microsystems Xenopus Express 2 Table of Contents General Information ........................................................................................................................... 5 Schedule of Events ............................................................................................................................. 6 Exhibitors ........................................................................................................................................... 8 Opening Session and Plenary/Platform Sessions ............................................................................ -

Inherited Human Sex Reversal Due to Impaired Nucleocytoplasmic
Inherited human sex reversal due to impaired PNAS PLUS nucleocytoplasmic trafficking of SRY defines a male transcriptional threshold Yen-Shan Chena, Joseph D. Raccaa, Nelson B. Phillipsa, and Michael A. Weissa,b,c,1 Departments of aBiochemistry, bBiomedical Engineering, and cMedicine, Case Western Reserve University, Cleveland, OH 44106 Edited by Patricia K. Donahoe, Massachusetts General Hospital, Boston, MA, and approved August 14, 2013 (received for review January 16, 2013) Human testis determination is initiated by SRY (sex determining studies of Swyer variants (6). Most often arising de novo in region on Y chromosome). Mutations in SRY cause gonadal dys- spermatogenesis and clustering in the HMG box (Fig. 1A), genesis with female somatic phenotype. Two subtle variants (V60L such mutations typically impair specific DNA binding by direct and I90M in the high-mobility group box) define inherited alleles or indirect perturbation of an angular protein–DNA interface shared by an XY sterile daughter and fertile father. Whereas spe- (6, 12). The present study focuses on inherited mutations cific DNA binding and bending are unaffected in a rat embryonic (V60L and I90M; asterisks in Fig. 1A) that by contrast allow pre-Sertoli cell line, the variants exhibited selective defects in near-native DNA binding and bending (13, 14). Such mutations are nucleocytoplasmic shuttling due to impaired nuclear import compatible with alternative developmental outcomes: testicular (V60L; mediated by Exportin-4) or export (I90M; mediated by chro- differentiation leading to virilization (fertile 46,XY father) or na- mosome region maintenance 1). Decreased shuttling limits nuclear scent ovarian differentiation leading to gonadal dysgenesis (sterile 46,XY daughter) (13).