Manuscrit De Thèse
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

A Computational Approach for Defining a Signature of Β-Cell Golgi Stress in Diabetes Mellitus
Page 1 of 781 Diabetes A Computational Approach for Defining a Signature of β-Cell Golgi Stress in Diabetes Mellitus Robert N. Bone1,6,7, Olufunmilola Oyebamiji2, Sayali Talware2, Sharmila Selvaraj2, Preethi Krishnan3,6, Farooq Syed1,6,7, Huanmei Wu2, Carmella Evans-Molina 1,3,4,5,6,7,8* Departments of 1Pediatrics, 3Medicine, 4Anatomy, Cell Biology & Physiology, 5Biochemistry & Molecular Biology, the 6Center for Diabetes & Metabolic Diseases, and the 7Herman B. Wells Center for Pediatric Research, Indiana University School of Medicine, Indianapolis, IN 46202; 2Department of BioHealth Informatics, Indiana University-Purdue University Indianapolis, Indianapolis, IN, 46202; 8Roudebush VA Medical Center, Indianapolis, IN 46202. *Corresponding Author(s): Carmella Evans-Molina, MD, PhD ([email protected]) Indiana University School of Medicine, 635 Barnhill Drive, MS 2031A, Indianapolis, IN 46202, Telephone: (317) 274-4145, Fax (317) 274-4107 Running Title: Golgi Stress Response in Diabetes Word Count: 4358 Number of Figures: 6 Keywords: Golgi apparatus stress, Islets, β cell, Type 1 diabetes, Type 2 diabetes 1 Diabetes Publish Ahead of Print, published online August 20, 2020 Diabetes Page 2 of 781 ABSTRACT The Golgi apparatus (GA) is an important site of insulin processing and granule maturation, but whether GA organelle dysfunction and GA stress are present in the diabetic β-cell has not been tested. We utilized an informatics-based approach to develop a transcriptional signature of β-cell GA stress using existing RNA sequencing and microarray datasets generated using human islets from donors with diabetes and islets where type 1(T1D) and type 2 diabetes (T2D) had been modeled ex vivo. To narrow our results to GA-specific genes, we applied a filter set of 1,030 genes accepted as GA associated. -
Supplementary Table 1
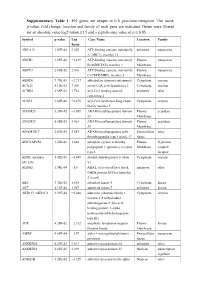
Supplementary Table 1. 492 genes are unique to 0 h post-heat timepoint. The name, p-value, fold change, location and family of each gene are indicated. Genes were filtered for an absolute value log2 ration 1.5 and a significance value of p ≤ 0.05. Symbol p-value Log Gene Name Location Family Ratio ABCA13 1.87E-02 3.292 ATP-binding cassette, sub-family unknown transporter A (ABC1), member 13 ABCB1 1.93E-02 −1.819 ATP-binding cassette, sub-family Plasma transporter B (MDR/TAP), member 1 Membrane ABCC3 2.83E-02 2.016 ATP-binding cassette, sub-family Plasma transporter C (CFTR/MRP), member 3 Membrane ABHD6 7.79E-03 −2.717 abhydrolase domain containing 6 Cytoplasm enzyme ACAT1 4.10E-02 3.009 acetyl-CoA acetyltransferase 1 Cytoplasm enzyme ACBD4 2.66E-03 1.722 acyl-CoA binding domain unknown other containing 4 ACSL5 1.86E-02 −2.876 acyl-CoA synthetase long-chain Cytoplasm enzyme family member 5 ADAM23 3.33E-02 −3.008 ADAM metallopeptidase domain Plasma peptidase 23 Membrane ADAM29 5.58E-03 3.463 ADAM metallopeptidase domain Plasma peptidase 29 Membrane ADAMTS17 2.67E-04 3.051 ADAM metallopeptidase with Extracellular other thrombospondin type 1 motif, 17 Space ADCYAP1R1 1.20E-02 1.848 adenylate cyclase activating Plasma G-protein polypeptide 1 (pituitary) receptor Membrane coupled type I receptor ADH6 (includes 4.02E-02 −1.845 alcohol dehydrogenase 6 (class Cytoplasm enzyme EG:130) V) AHSA2 1.54E-04 −1.6 AHA1, activator of heat shock unknown other 90kDa protein ATPase homolog 2 (yeast) AK5 3.32E-02 1.658 adenylate kinase 5 Cytoplasm kinase AK7 -

Cells Receptor-Evoked Responses of Human Mast RGS13 Controls G
RGS13 Controls G Protein-Coupled Receptor-Evoked Responses of Human Mast Cells This information is current as Geetanjali Bansal, Jeffrey A. DiVietro, Hye Sun Kuehn, of September 25, 2021. Sudhir Rao, Karl H. Nocka, Alasdair M. Gilfillan and Kirk M. Druey J Immunol 2008; 181:7882-7890; ; doi: 10.4049/jimmunol.181.11.7882 http://www.jimmunol.org/content/181/11/7882 Downloaded from References This article cites 65 articles, 27 of which you can access for free at: http://www.jimmunol.org/content/181/11/7882.full#ref-list-1 http://www.jimmunol.org/ Why The JI? Submit online. • Rapid Reviews! 30 days* from submission to initial decision • No Triage! Every submission reviewed by practicing scientists • Fast Publication! 4 weeks from acceptance to publication by guest on September 25, 2021 *average Subscription Information about subscribing to The Journal of Immunology is online at: http://jimmunol.org/subscription Permissions Submit copyright permission requests at: http://www.aai.org/About/Publications/JI/copyright.html Email Alerts Receive free email-alerts when new articles cite this article. Sign up at: http://jimmunol.org/alerts The Journal of Immunology is published twice each month by The American Association of Immunologists, Inc., 1451 Rockville Pike, Suite 650, Rockville, MD 20852 Copyright © 2008 by The American Association of Immunologists All rights reserved. Print ISSN: 0022-1767 Online ISSN: 1550-6606. The Journal of Immunology RGS13 Controls G Protein-Coupled Receptor-Evoked Responses of Human Mast Cells1 Geetanjali Bansal,2* Jeffrey A. DiVietro,* Hye Sun Kuehn,† Sudhir Rao,3‡ Karl H. Nocka,4‡ Alasdair M. Gilfillan,4† and Kirk M. -

Systems Vaccinology for a Live Attenuated Tularemia Vaccine Reveals Unique Transcriptional Signatures That Predict Humoral and Cellular Immune Responses"’ Version 1.0
Supplementary Text Manuscript Appendix "‘Systems vaccinology for a live attenuated tularemia vaccine reveals unique transcriptional signatures that predict humoral and cellular immune responses"’ Version 1.0 December 23, 2019 Table of Contents 1 Introduction 8 2 Supplemental methods 8 2.1 Study Design . 8 2.2 Microarray experiment . 8 2.3 Cell mediated immunity experiments . 9 2.4 Microarray data preprocessing . 10 2.5 Differential gene analysis . 11 2.6 Determination of robust gene clusters . 11 2.7 Gene set enrichment analysis . 11 2.8 Regularized logistic regression analysis . 12 2.9 Regularized canonical correlation analysis . 13 2.10 Comparisons with viral vaccine microarray studies . 14 2.11 Cell mediated immunity analysis . 15 3 Supplemental results 15 4 References 84 1 List of Tables Table 1 Descriptive summary statistics of percent monocyte cells of live PBMCs fold change from baseline by treatment. 16 Table 2 Descriptive summary statistics of percent monocyte cells (CD16+) of live PBMCs fold change from baseline by treatment. 16 Table 3 Descriptive summary statistics of percent monocyte cells (CD16-) of live PBMCs fold change from baseline by treatment. 16 Table 4 Descriptive summary statistics of percent T-cells of live PBMCs fold change from baseline by treatment. 16 Table 5 Descriptive summary statistics of percent T-cells (CD4+) of live PBMCs fold change from baseline by treatment. 16 Table 6 Descriptive summary statistics of percent T-cells (CD4-) of live PBMCs fold change from baseline by treatment. 17 Table 7 Descriptive summary statistics of percent natural killer cells (CD56 bright) of live PBMCs fold change from baseline by treatment. -

Function and Regulation of Heterotrimeric G Proteins During Chemotaxis Kamp, Marjon E; Liu, Youtao; Kortholt, Arjan
University of Groningen Function and Regulation of Heterotrimeric G Proteins during Chemotaxis Kamp, Marjon E; Liu, Youtao; Kortholt, Arjan Published in: International Journal of Molecular Sciences DOI: 10.3390/ijms17010090 IMPORTANT NOTE: You are advised to consult the publisher's version (publisher's PDF) if you wish to cite from it. Please check the document version below. Document Version Publisher's PDF, also known as Version of record Publication date: 2016 Link to publication in University of Groningen/UMCG research database Citation for published version (APA): Kamp, M. E., Liu, Y., & Kortholt, A. (2016). Function and Regulation of Heterotrimeric G Proteins during Chemotaxis. International Journal of Molecular Sciences, 17(1), [90]. https://doi.org/10.3390/ijms17010090 Copyright Other than for strictly personal use, it is not permitted to download or to forward/distribute the text or part of it without the consent of the author(s) and/or copyright holder(s), unless the work is under an open content license (like Creative Commons). Take-down policy If you believe that this document breaches copyright please contact us providing details, and we will remove access to the work immediately and investigate your claim. Downloaded from the University of Groningen/UMCG research database (Pure): http://www.rug.nl/research/portal. For technical reasons the number of authors shown on this cover page is limited to 10 maximum. Download date: 26-09-2021 International Journal of Molecular Sciences Review Function and Regulation of Heterotrimeric G Proteins during Chemotaxis Marjon E. Kamp †, Youtao Liu † and Arjan Kortholt * Received: 28 November 2015; Accepted: 31 December 2015; Published: 14 January 2016 Academic Editor: Kathleen Van Craenenbroeck Department of Cell Biochemistry, University of Groningen, Nijenborgh 7, 9747 AG Groningen, The Netherlands; [email protected] (M.E.K.); [email protected] (Y.L.) * Correspondence: [email protected]; Tel.: +31-50-363-4206 † These authors contributed equally to this work. -

Rgs13 Constrains Early B Cell Responses and Limits Germinal Center Sizes
Rgs13 Constrains Early B Cell Responses and Limits Germinal Center Sizes Il-Young Hwang, Kyung-Sun Hwang, Chung Park, Kathleen A. Harrison, John H. Kehrl* B Cell Molecular Immunology Section, Laboratory of Immunoregulation, National Institute of Allergy and Infectious Diseases, National Institutes of Health, Bethesda, Maryland, United States of America Abstract Germinal centers (GCs) are microanatomic structures that develop in secondary lymphoid organs in response to antigenic stimulation. Within GCs B cells clonally expand and their immunoglobulin genes undergo class switch recombination and somatic hypermutation. Transcriptional profiling has identified a number of genes that are prominently expressed in GC B cells. Among them is Rgs13, which encodes an RGS protein with a dual function. Its canonical function is to accelerate the intrinsic GTPase activity of heterotrimeric G-protein a subunits at the plasma membrane, thereby limiting heterotrimeric G- protein signaling. A unique, non-canonical function of RGS13 occurs following translocation to the nucleus, where it represses CREB transcriptional activity. The functional role of RGS13 in GC B cells is unknown. To create a surrogate marker for Rgs13 expression and a loss of function mutation, we inserted a GFP coding region into the Rgs13 genomic locus. Following immunization GFP expression rapidly increased in activated B cells, persisted in GC B cells, but declined in newly generated memory B and plasma cells. Intravital microscopy of the inguinal lymph node (LN) of immunized mice revealed the rapid appearance of GFP+ cells at LN interfollicular regions and along the T/B cell borders, and eventually within GCs. Analysis of WT, knock-in, and mixed chimeric mice indicated that RGS13 constrains extra-follicular plasma cell generation, GC size, and GC B cell numbers. -

Flavone Effects on the Proteome and Transcriptome of Colonocytes in Vitro and in Vivo and Its Relevance for Cancer Prevention and Therapy
TECHNISCHE UNIVERSITÄT MÜNCHEN Lehrstuhl für Ernährungsphysiologie Flavone effects on the proteome and transcriptome of colonocytes in vitro and in vivo and its relevance for cancer prevention and therapy Isabel Winkelmann Vollständiger Abdruck der von der Fakultät Wissenschaftszentrum Weihenstephan für Ernährung, Landnutzung und Umwelt der Technischen Universität München zur Erlangung des akademischen Grades eines Doktors der Naturwissenschaften genehmigten Dissertation. Vorsitzender: Univ.-Prof. Dr. D. Haller Prüfer der Dissertation: 1. Univ.-Prof. Dr. H. Daniel 2. Univ.-Prof. Dr. U. Wenzel (Justus-Liebig-Universität Giessen) 3. Prof. Dr. E.C.M. Mariman (Maastricht University, Niederlande) schriftliche Beurteilung Die Dissertation wurde am 24.08.2009 bei der Technischen Universität München eingereicht und durch die Fakultät Wissenschaftszentrum Weihenstephan für Ernährung, Landnutzung und Umwelt am 25.11.2009 angenommen. Die Forschung ist immer auf dem Wege, nie am Ziel. (Adolf Pichler) Table of contents 1. Introduction .......................................................................................................... 1 1.1. Cancer and carcinogenesis .................................................................................. 2 1.2. Colorectal Cancer ............................................................................................... 3 1.2.1. Hereditary forms of CRC ........................................................................................ 4 1.2.2. Sporadic forms of CRC .......................................................................................... -

AKT-Mtor Signaling in Human Acute Myeloid Leukemia Cells and Its Association with Adverse Prognosis
Cancers 2018, 10, 332 S1 of S35 Supplementary Materials: Clonal Heterogeneity Reflected by PI3K- AKT-mTOR Signaling in Human Acute Myeloid Leukemia Cells and its Association with Adverse Prognosis Ina Nepstad, Kimberley Joanne Hatfield, Tor Henrik Anderson Tvedt, Håkon Reikvam and Øystein Bruserud Figure S1. Detection of clonal heterogeneity for 49 acute myeloid leukemia (AML) patients; the results from representative flow cytometric analyses of phosphatidylinositol-3-kinase-Akt-mechanistic target of rapamycin (PI3K-Akt-mTOR) activation. For each patient clonal heterogeneity was detected by analysis Cancers 2018, 10, 332 S2 of S35 of at least one mediator in the PI3K-Akt-mTOR pathway. Patient ID is shown in the upper right corner of each histogram. The figure documents the detection of dual populations for all patients, showing the results from one representative flow cytometric analysis for each of these 49 patients. The Y-axis represents the amount of cells, and the X-axis represents the fluorescence intensity. The stippled line shows the negative/unstained controls. Figure S2. Cell preparation and gating strategy. Flow cytometry was used for examination of the constitutive expression of the mediators in the PI3K-Akt-mTOR pathway/network in primary AML cells. Cryopreserved cells were thawed and washed before suspension cultures were prepared as described in Materials and methods. Briefly, cryopreserved and thawed primary leukemic cells were incubated for 20 minutes in RPMI-1640 (Sigma-Aldrich) before being directly fixed in 1.5% paraformaldehyde (PFA) and permeabilized with 100% ice-cold methanol. The cells were thereafter rehydrated by adding 2 mL phosphate buffered saline (PBS), gently re-suspended and then centrifuged. -

RGS13 Goat Polyclonal Antibody – TA305720 | Origene
OriGene Technologies, Inc. 9620 Medical Center Drive, Ste 200 Rockville, MD 20850, US Phone: +1-888-267-4436 [email protected] EU: [email protected] CN: [email protected] Product datasheet for TA305720 RGS13 Goat Polyclonal Antibody Product data: Product Type: Primary Antibodies Applications: WB Recommended Dilution: WB: 0.5-1.5ug/ml. Reactivity: Human Host: Goat Isotype: IgG Clonality: Polyclonal Immunogen: Peptide with sequence DESKRPPSNLTLEE-C, from the N Terminus of the protein sequence according to NP_658912.1. Formulation: 0.5 mg/ml in Tris saline, 0.02% sodium azide, pH7.3 with 0.5% bovine serum albumin Concentration: lot specific Purification: Purified from goat serum by ammonium sulphate precipitation followed by antigen affinity chromatography using the immunizing peptide. Supplied at 0.5 mg/ml in Tris saline, 0.02% sodium azide, pH7.3 with 0.5% bovine serum albumin. Aliquot and store at -20C. Minimize freezing and thawing. Conjugation: Unconjugated Storage: Store at -20°C as received. Stability: Stable for 12 months from date of receipt. Gene Name: regulator of G-protein signaling 13 Database Link: NP_002918 Entrez Gene 6003 Human O14921 This product is to be used for laboratory only. Not for diagnostic or therapeutic use. View online » ©2021 OriGene Technologies, Inc., 9620 Medical Center Drive, Ste 200, Rockville, MD 20850, US 1 / 2 RGS13 Goat Polyclonal Antibody – TA305720 Background: The protein encoded by this gene is a member of the regulator of G protein signaling (RGS) family. RGS family members share similarity with S. cerevisiae SST2 and C. elegans egl-10 proteins, which contain a characteristic conserved RGS domain. -

A Systems Immunology Approach Identifies the Collective Impact of Five Mirs in Th2 Inflammation Supplementary Information
A systems immunology approach identifies the collective impact of five miRs in Th2 inflammation Supplementary information Ayşe Kılıç, Marc Santolini, Taiji Nakano, Matthias Schiller, Mizue Teranishi,Pascal Gellert, Yuliya Ponomareva, Thomas Braun, Shizuka Uchida, Scott T. Weiss, Amitabh Sharma and Harald Renz Corresponding Author: Harald Renz, MD Institute of Laboratory Medicine Philipps University Marburg 35043 Marburg, Germany Phone +49 6421 586 6235 [email protected] 1 Kılıç A et. al. 2018 A B C D PBS OVA chronic PAS PAS (cm H2O*s/ml (cm Raw Sirius Red Sirius Supplementary figure 1: Characteristic phenotype of the acute and chronic allergic airway inflammatory response in the OVA-induced mouse model. (a) Total and differential cell counts determined in BAL and cytospins show a dominant influx of eosinophils. (b) Serum titers of OVA-specific immunoglobulins are elevated. (c) Representative measurement of airway reactivity to increasing methacholine (Mch) responsiveness measured by head-out body plethysmography. (d) Representative lung section staining depicting airway inflammation and mucus production (PAS, original magnification x 10) and airway remodeling (SiriusRed; original magnification x 20). Data are presented as mean ± SEM and are representative for at least 3 independent experiments with n=8-10 animals per group. Statistical analyses have been performed with one-way ANOVA and Tukey’s post-test and show *p<0.05, **p<0.01 and ***p<0.001. 2 Kılıç A et. al. 2018 lymphocytes A lung activated CD4+-cells CD4+-T-cell-subpopulations SSC FSC ST2 live CD69 CD4 CXCR3 B spleen Naive CD4+- T cells DAPI FSC single cells SSC CD62L CD4 CD44 FSC FSC-W Supplementary figure 2. -

Supplementary Table S2. Cpuorfs Extracted from D
Supplementary Table S2. CPuORFs extracted from D. melanogaster , D. rerio , G. gallus , and H. sapiens . K a/K s analysis K a/K s analysis before manual validation after manual validation Gene † uORF-mORF Previous Species Gene ID Gene description Median Median CPuORF amino acid sequence symbol fusion ratio U -test U -test report pairwise q value pairwise HG number p value p value K a/K s ratio K a/K s ratio D. melanogaster FBgn0024734 PRL-1 PRL-1 0.00 0.06 0.0E+00 0.0E+00 0.06 0.0E+00 Crowe MCMAIVLVRPVPTSMEFHLDSAEYCQAQHK* ENSDARG00000006242 ptp4a1 protein tyrosine phosphatase type IVA, member 1 0.04 0.10 0.0E+00 0.0E+00 0.10 0.0E+00 Crowe MIKLQSSMSKHIPFCGVLGHTFMEFLKGSGDYCQAQHGVYAEK* ENSDARG00000035676 ptp4a2b protein tyrosine phosphatase type IVA, member 2b 0.00 0.22 0.0E+00 0.0E+00 0.22 0.0E+00 Crowe MVGPFFCFRVDSDHTYMAFVTVCREFCQAQHLTFADK* D. rerio ENSDARG00000039997 ptp4a3 protein tyrosine phosphatase type IVA, member 3 0.00 0.07 0.0E+00 0.0E+00 0.07 0.0E+00 Crowe MAFLQRTGDYCHAQHLHI* ENSDARG00000054814 PTP4A3 protein tyrosine phosphatase 4A3b 0.00 0.08 0.0E+00 0.0E+00 0.08 0.0E+00 Crowe MEFWQGSGDYCHAQHHTA* HG0001 ENSDARG00000087443 ptp4a2a protein tyrosine phosphatase type IVA, member 2a 0.00 0.14 0.0E+00 0.0E+00 0.14 0.0E+00 Crowe MQSSQLVFCSLIEGCTFMAFVSVSGDFCQAQHLVIVDK* ENSGALG00000003265 PTP4A2 protein tyrosine phosphatase type IVA, member 2 0.00 0.24 0.0E+00 0.0E+00 0.24 0.0E+00 Crowe MPGVFCWNLRRTFMAILSVRAEFCQAQHSALADK* G. gallus ENSGALG00000016271 PTP4A1 protein tyrosine phosphatase type IVA, member 1 0.00 0.08 0.0E+00 0.0E+00 0.08 0.0E+00 Crowe MIRLQSSMSNHIPQFCGVLGHTFMEFLKGSGDYCQAQHDLYADK* ENSG00000112245 PTP4A1 protein tyrosine phosphatase type IVA, member 1 0.00 0.09 0.0E+00 0.0E+00 0.09 0.0E+00 Crowe MIRPQSSMSKHIPQFCGVLGHTFMEFLKGSGDYCQAQHDLYADK* H. -

G Protein-Coupled Receptor Systems As Crucial Regulators of DNA Damage Response Processes
International Journal of Molecular Sciences Review G Protein-Coupled Receptor Systems as Crucial Regulators of DNA Damage Response Processes Hanne Leysen 1, Jaana van Gastel 1,2, Jhana O. Hendrickx 1,2 , Paula Santos-Otte 3, Bronwen Martin 1 and Stuart Maudsley 1,2,* 1 Department of Biomedical Sciences, University of Antwerp, 2610 Antwerp, Belgium; [email protected] (H.L.); [email protected] (J.v.G.); [email protected] (J.O.H.); [email protected] (B.M.) 2 Translational Neurobiology Group, Center of Molecular Neurology, VIB, 2610 Antwerp, Belgium 3 Institute of Biophysics, Humboldt-Universität zu Berlin, 10115 Berlin, Germany; [email protected] * Correspondence: [email protected]; Tel.: +32-3265-1057 Received: 22 August 2018; Accepted: 15 September 2018; Published: 26 September 2018 Abstract: G protein-coupled receptors (GPCRs) and their associated proteins represent one of the most diverse cellular signaling systems involved in both physiological and pathophysiological processes. Aging represents perhaps the most complex biological process in humans and involves a progressive degradation of systemic integrity and physiological resilience. This is in part mediated by age-related aberrations in energy metabolism, mitochondrial function, protein folding and sorting, inflammatory activity and genomic stability. Indeed, an increased rate of unrepaired DNA damage is considered to be one of the ‘hallmarks’ of aging. Over the last two decades our appreciation of the complexity of GPCR signaling systems has expanded their functional signaling repertoire. One such example of this is the incipient role of GPCRs and GPCR-interacting proteins in DNA damage and repair mechanisms.