Circadian Modulation of Genome-Wide Rxr Binding in Mouse Liver Tissue
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

Genome-Wide Approach to Identify Risk Factors for Therapy-Related Myeloid Leukemia
Leukemia (2006) 20, 239–246 & 2006 Nature Publishing Group All rights reserved 0887-6924/06 $30.00 www.nature.com/leu ORIGINAL ARTICLE Genome-wide approach to identify risk factors for therapy-related myeloid leukemia A Bogni1, C Cheng2, W Liu2, W Yang1, J Pfeffer1, S Mukatira3, D French1, JR Downing4, C-H Pui4,5,6 and MV Relling1,6 1Department of Pharmaceutical Sciences, The University of Tennessee, Memphis, TN, USA; 2Department of Biostatistics, The University of Tennessee, Memphis, TN, USA; 3Hartwell Center, The University of Tennessee, Memphis, TN, USA; 4Department of Pathology, The University of Tennessee, Memphis, TN, USA; 5Department of Hematology/Oncology St Jude Children’s Research Hospital, The University of Tennessee, Memphis, TN, USA; and 6Colleges of Medicine and Pharmacy, The University of Tennessee, Memphis, TN, USA Using a target gene approach, only a few host genetic risk therapy increases, the importance of identifying host factors for factors for treatment-related myeloid leukemia (t-ML) have been secondary neoplasms increases. defined. Gene expression microarrays allow for a more 4 genome-wide approach to assess possible genetic risk factors Because DNA microarrays interrogate multiple ( 10 000) for t-ML. We assessed gene expression profiles (n ¼ 12 625 genes in one experiment, they allow for a ‘genome-wide’ probe sets) in diagnostic acute lymphoblastic leukemic cells assessment of genes that may predispose to leukemogenesis. from 228 children treated on protocols that included leukemo- DNA microarray analysis of gene expression has been used to genic agents such as etoposide, 13 of whom developed t-ML. identify distinct expression profiles that are characteristic of Expression of 68 probes, corresponding to 63 genes, was different leukemia subtypes.13,14 Studies using this method have significantly related to risk of t-ML. -

Human Retinoic Acid Signaling
RT² Profiler PCR Array (96-Well Format and 384-Well [4 x 96] Format) Human Retinoic Acid Signaling Cat. no. 330231 PAHS-180Z For pathway expression analysis Format For use with the following real-time cyclers RT² Profiler PCR Array, Applied Biosystems® models 5700, 7000, 7300, 7500, Format A 7700, 7900HT, ViiA™ 7 (96-well block); Bio-Rad® models iCycler®, iQ™5, MyiQ™, MyiQ2; Bio-Rad/MJ Research Chromo4™; Eppendorf® Mastercycler® ep realplex models 2, 2s, 4, 4s; Stratagene® models Mx3005P®, Mx3000P®; Takara TP-800 RT² Profiler PCR Array, Applied Biosystems models 7500 (Fast block), 7900HT (Fast Format C block), StepOnePlus™, ViiA 7 (Fast block) RT² Profiler PCR Array, Bio-Rad CFX96™; Bio-Rad/MJ Research models DNA Format D Engine Opticon®, DNA Engine Opticon 2; Stratagene Mx4000® RT² Profiler PCR Array, Applied Biosystems models 7900HT (384-well block), ViiA 7 Format E (384-well block); Bio-Rad CFX384™ RT² Profiler PCR Array, Roche® LightCycler® 480 (96-well block) Format F RT² Profiler PCR Array, Roche LightCycler 480 (384-well block) Format G RT² Profiler PCR Array, Fluidigm® BioMark™ Format H Sample & Assay Technologies Description The Human Retinoic Acid Pathway RT² Profiler PCR Array profiles the expression of 84 key genes involved in retinoic acid signaling. Retinoic acid (RA) is the primary functional derivative of vitamin A (retinol) and its activity is implicated in many aspects of vertebrate development and homeostasis, while disruptions in this pathway cause developmental abnormalities and disrupt function in adipose, cardiac, nervous, reproductive, and integumentary tissues, among others. RA acts primarily by binding a family of nuclear receptors (the retinoic acid receptors alpha, beta, and gamma) that then heterodimerize with their partners (the retinoid X receptors alpha, beta, and gamma) and alter transcription. -

Role of Nuclear Receptors in Central Nervous System Development and Associated Diseases
Role of Nuclear Receptors in Central Nervous System Development and Associated Diseases The Harvard community has made this article openly available. Please share how this access benefits you. Your story matters Citation Olivares, Ana Maria, Oscar Andrés Moreno-Ramos, and Neena B. Haider. 2015. “Role of Nuclear Receptors in Central Nervous System Development and Associated Diseases.” Journal of Experimental Neuroscience 9 (Suppl 2): 93-121. doi:10.4137/JEN.S25480. http:// dx.doi.org/10.4137/JEN.S25480. Published Version doi:10.4137/JEN.S25480 Citable link http://nrs.harvard.edu/urn-3:HUL.InstRepos:27320246 Terms of Use This article was downloaded from Harvard University’s DASH repository, and is made available under the terms and conditions applicable to Other Posted Material, as set forth at http:// nrs.harvard.edu/urn-3:HUL.InstRepos:dash.current.terms-of- use#LAA Journal name: Journal of Experimental Neuroscience Journal type: Review Year: 2015 Volume: 9(S2) Role of Nuclear Receptors in Central Nervous System Running head verso: Olivares et al Development and Associated Diseases Running head recto: Nuclear receptors development and associated diseases Supplementary Issue: Molecular and Cellular Mechanisms of Neurodegeneration Ana Maria Olivares1, Oscar Andrés Moreno-Ramos2 and Neena B. Haider1 1Department of Ophthalmology, Schepens Eye Research Institute, Massachusetts Eye and Ear, Harvard Medical School, Boston, MA, USA. 2Departamento de Ciencias Biológicas, Facultad de Ciencias, Universidad de los Andes, Bogotá, Colombia. ABSTRACT: The nuclear hormone receptor (NHR) superfamily is composed of a wide range of receptors involved in a myriad of important biological processes, including development, growth, metabolism, and maintenance. -

Supplementary Table S4. FGA Co-Expressed Gene List in LUAD
Supplementary Table S4. FGA co-expressed gene list in LUAD tumors Symbol R Locus Description FGG 0.919 4q28 fibrinogen gamma chain FGL1 0.635 8p22 fibrinogen-like 1 SLC7A2 0.536 8p22 solute carrier family 7 (cationic amino acid transporter, y+ system), member 2 DUSP4 0.521 8p12-p11 dual specificity phosphatase 4 HAL 0.51 12q22-q24.1histidine ammonia-lyase PDE4D 0.499 5q12 phosphodiesterase 4D, cAMP-specific FURIN 0.497 15q26.1 furin (paired basic amino acid cleaving enzyme) CPS1 0.49 2q35 carbamoyl-phosphate synthase 1, mitochondrial TESC 0.478 12q24.22 tescalcin INHA 0.465 2q35 inhibin, alpha S100P 0.461 4p16 S100 calcium binding protein P VPS37A 0.447 8p22 vacuolar protein sorting 37 homolog A (S. cerevisiae) SLC16A14 0.447 2q36.3 solute carrier family 16, member 14 PPARGC1A 0.443 4p15.1 peroxisome proliferator-activated receptor gamma, coactivator 1 alpha SIK1 0.435 21q22.3 salt-inducible kinase 1 IRS2 0.434 13q34 insulin receptor substrate 2 RND1 0.433 12q12 Rho family GTPase 1 HGD 0.433 3q13.33 homogentisate 1,2-dioxygenase PTP4A1 0.432 6q12 protein tyrosine phosphatase type IVA, member 1 C8orf4 0.428 8p11.2 chromosome 8 open reading frame 4 DDC 0.427 7p12.2 dopa decarboxylase (aromatic L-amino acid decarboxylase) TACC2 0.427 10q26 transforming, acidic coiled-coil containing protein 2 MUC13 0.422 3q21.2 mucin 13, cell surface associated C5 0.412 9q33-q34 complement component 5 NR4A2 0.412 2q22-q23 nuclear receptor subfamily 4, group A, member 2 EYS 0.411 6q12 eyes shut homolog (Drosophila) GPX2 0.406 14q24.1 glutathione peroxidase -
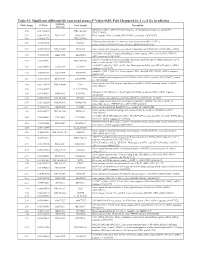
(P -Value<0.05, Fold Change≥1.4), 4 Vs. 0 Gy Irradiation
Table S1: Significant differentially expressed genes (P -Value<0.05, Fold Change≥1.4), 4 vs. 0 Gy irradiation Genbank Fold Change P -Value Gene Symbol Description Accession Q9F8M7_CARHY (Q9F8M7) DTDP-glucose 4,6-dehydratase (Fragment), partial (9%) 6.70 0.017399678 THC2699065 [THC2719287] 5.53 0.003379195 BC013657 BC013657 Homo sapiens cDNA clone IMAGE:4152983, partial cds. [BC013657] 5.10 0.024641735 THC2750781 Ciliary dynein heavy chain 5 (Axonemal beta dynein heavy chain 5) (HL1). 4.07 0.04353262 DNAH5 [Source:Uniprot/SWISSPROT;Acc:Q8TE73] [ENST00000382416] 3.81 0.002855909 NM_145263 SPATA18 Homo sapiens spermatogenesis associated 18 homolog (rat) (SPATA18), mRNA [NM_145263] AA418814 zw01a02.s1 Soares_NhHMPu_S1 Homo sapiens cDNA clone IMAGE:767978 3', 3.69 0.03203913 AA418814 AA418814 mRNA sequence [AA418814] AL356953 leucine-rich repeat-containing G protein-coupled receptor 6 {Homo sapiens} (exp=0; 3.63 0.0277936 THC2705989 wgp=1; cg=0), partial (4%) [THC2752981] AA484677 ne64a07.s1 NCI_CGAP_Alv1 Homo sapiens cDNA clone IMAGE:909012, mRNA 3.63 0.027098073 AA484677 AA484677 sequence [AA484677] oe06h09.s1 NCI_CGAP_Ov2 Homo sapiens cDNA clone IMAGE:1385153, mRNA sequence 3.48 0.04468495 AA837799 AA837799 [AA837799] Homo sapiens hypothetical protein LOC340109, mRNA (cDNA clone IMAGE:5578073), partial 3.27 0.031178378 BC039509 LOC643401 cds. [BC039509] Homo sapiens Fas (TNF receptor superfamily, member 6) (FAS), transcript variant 1, mRNA 3.24 0.022156298 NM_000043 FAS [NM_000043] 3.20 0.021043295 A_32_P125056 BF803942 CM2-CI0135-021100-477-g08 CI0135 Homo sapiens cDNA, mRNA sequence 3.04 0.043389246 BF803942 BF803942 [BF803942] 3.03 0.002430239 NM_015920 RPS27L Homo sapiens ribosomal protein S27-like (RPS27L), mRNA [NM_015920] Homo sapiens tumor necrosis factor receptor superfamily, member 10c, decoy without an 2.98 0.021202829 NM_003841 TNFRSF10C intracellular domain (TNFRSF10C), mRNA [NM_003841] 2.97 0.03243901 AB002384 C6orf32 Homo sapiens mRNA for KIAA0386 gene, partial cds. -

Interplay Between P53 and Epigenetic Pathways in Cancer
University of Pennsylvania ScholarlyCommons Publicly Accessible Penn Dissertations 2016 Interplay Between P53 and Epigenetic Pathways in Cancer Jiajun Zhu University of Pennsylvania, [email protected] Follow this and additional works at: https://repository.upenn.edu/edissertations Part of the Biology Commons, Cell Biology Commons, and the Molecular Biology Commons Recommended Citation Zhu, Jiajun, "Interplay Between P53 and Epigenetic Pathways in Cancer" (2016). Publicly Accessible Penn Dissertations. 2130. https://repository.upenn.edu/edissertations/2130 This paper is posted at ScholarlyCommons. https://repository.upenn.edu/edissertations/2130 For more information, please contact [email protected]. Interplay Between P53 and Epigenetic Pathways in Cancer Abstract The human TP53 gene encodes the most potent tumor suppressor protein p53. More than half of all human cancers contain mutations in the TP53 gene, while the majority of the remaining cases involve other mechanisms to inactivate wild-type p53 function. In the first part of my dissertation research, I have explored the mechanism of suppressed wild-type p53 activity in teratocarcinoma. In the teratocarcinoma cell line NTera2, we show that wild-type p53 is mono-methylated at Lysine 370 and Lysine 382. These post-translational modifications contribute ot the compromised tumor suppressive activity of p53 despite a high level of wild-type protein in NTera2 cells. This study provides evidence for an epigenetic mechanism that cancer cells can exploit to inactivate p53 wild-type function. The paradigm provides insight into understanding the modes of p53 regulation, and can likely be applied to other cancer types with wild-type p53 proteins. On the other hand, cancers with TP53 mutations are mostly found to contain missense substitutions of the TP53 gene, resulting in expression of full length, but mutant forms of p53 that confer tumor-promoting “gain-of-function” (GOF) to cancer. -

Alternative Splicing in the Nuclear Receptor Superfamily Expands Gene Function to Refine Endo-Xenobiotic Metabolism S
Supplemental material to this article can be found at: http://dmd.aspetjournals.org/content/suppl/2020/01/24/dmd.119.089102.DC1 1521-009X/48/4/272–287$35.00 https://doi.org/10.1124/dmd.119.089102 DRUG METABOLISM AND DISPOSITION Drug Metab Dispos 48:272–287, April 2020 Copyright ª 2020 by The American Society for Pharmacology and Experimental Therapeutics Minireview Alternative Splicing in the Nuclear Receptor Superfamily Expands Gene Function to Refine Endo-Xenobiotic Metabolism s Andrew J. Annalora, Craig B. Marcus, and Patrick L. Iversen Department of Environmental and Molecular Toxicology, Oregon State University, Corvallis, Oregon (A.J.A., C.B.M., P.L.I.) and United States Army Research Institute for Infectious Disease, Frederick, Maryland (P.L.I.) Received August 16, 2019; accepted December 31, 2019 ABSTRACT Downloaded from The human genome encodes 48 nuclear receptor (NR) genes, whose Exon inclusion options are differentially distributed across NR translated products transform chemical signals from endo- subfamilies, suggesting group-specific conservation of resilient func- xenobiotics into pleotropic RNA transcriptional profiles that refine tionalities. A deeper understanding of this transcriptional plasticity drug metabolism. This review describes the remarkable diversifica- expands our understanding of how chemical signals are refined and tion of the 48 human NR genes, which are potentially processed into mediated by NR genes. This expanded view of the NR transcriptome over 1000 distinct mRNA transcripts by alternative splicing (AS). The informs new models of chemical toxicity, disease diagnostics, and dmd.aspetjournals.org average human NR expresses ∼21 transcripts per gene and is precision-based approaches to personalized medicine. -

Epigenomic and Transcriptional Regulation of Hepatic Metabolism by REV-ERB and Hdac3
University of Pennsylvania ScholarlyCommons Publicly Accessible Penn Dissertations 2013 Epigenomic and Transcriptional Regulation of Hepatic Metabolism by REV-ERB and Hdac3 Dan Feng University of Pennsylvania, [email protected] Follow this and additional works at: https://repository.upenn.edu/edissertations Part of the Genetics Commons, and the Molecular Biology Commons Recommended Citation Feng, Dan, "Epigenomic and Transcriptional Regulation of Hepatic Metabolism by REV-ERB and Hdac3" (2013). Publicly Accessible Penn Dissertations. 633. https://repository.upenn.edu/edissertations/633 This paper is posted at ScholarlyCommons. https://repository.upenn.edu/edissertations/633 For more information, please contact [email protected]. Epigenomic and Transcriptional Regulation of Hepatic Metabolism by REV-ERB and Hdac3 Abstract Metabolic activities are regulated by the circadian clock, and disruption of the clock exacerbates metabolic diseases including obesity and diabetes. Transcriptomic studies in metabolic organs suggested that the circadian clock drives the circadian expression of important metabolic genes. Here we show that histone deacetylase 3 (HDAC3) is recruited to the mouse liver genome in a circadian manner. Histone acetylation is inversely related to HDAC3 binding, and this rhythm is lost when HDAC3 is absent. Diurnal recruitment of HDAC3 corresponds to the expression pattern of REV-ERBα, an important component of the circadian clock. REV-ERBα colocalizes with HDAC3 near genes regulating lipid metabolism, and deletion of HDAC3 or Rev-erbα in mouse liver causes hepatic steatosis. Thus, genomic recruitment of HDAC3 by REV-ERBα directs a circadian rhythm of histone acetylation and gene expression required for normal hepatic lipid homeostasis. In addition, we reported that the REV-ERBα paralog, REV- ERBβ also displays circadian binding similar to that of REV-ERBα. -

Transcriptional Regulation by V-Erba, the Oncogenic Counterpart of Thyroid Hormone Receptor (TR)
PDF hosted at the Radboud Repository of the Radboud University Nijmegen The following full text is a publisher's version. For additional information about this publication click this link. http://hdl.handle.net/2066/19007 Please be advised that this information was generated on 2021-10-04 and may be subject to change. Transcriptional Regulation by v-ErbA, the Oncogenic Counterpart of Thyroid Hormone Receptor (TR) Doctoral dissertation to obtain the degree of doctor from the University of Nijmegen, The Netherlands according to the decision of the Council of Deans to be defended in public on Tuesday the 23rd of January 2001 at 13.30 by Georgia Braliou Supervisor: Prof. Dr. Ir. H.G. Stunnenberg Manuscript committee: Prof. H.P.J. Bloemers Prof. Dr. J.A. Schalken Dr. M. von Lindern (Erasmus University, Rotterdam) Chapter 2 is reproduced by permission of Oxford University Press Chapter 3 is reproduced by permission of Nature Publishing Group Print Partners IPSKAMP, Enschede W emeyalwOeig ta epga Sou Kopie! nanta en sofia en oih oaç Yalm 104 (103) :24 O Lord, how magnified are thy works! In wisdom have thou made them all Psalm 104 (103):24 Contens Abbreviations 9 Chapter 1 Introduction 11 Chapter 2 Leukemic transformation by the v-ErbA oncoprotein 75 entails constitutive binding to and repression of an erythroid enhancer in vivo Chapter 3 The v-ErbA oncoprotein quenches the activity of an 111 erythroid-specific enhancer Chapter 4 The C-terminus of NCoR interacts with v-ErbA in 141 AEV transformed cells but does not abrogate the repressive activity -

RAB-GAP Immunity Signaling by the Tbc1d23 Spatiotemporal Inhibition
Downloaded from http://www.jimmunol.org/ by guest on September 27, 2021 is online at: average * The Journal of Immunology , 17 of which you can access for free at: 2012; 188:2905-2913; Prepublished online 6 from submission to initial decision 4 weeks from acceptance to publication February 2012; doi: 10.4049/jimmunol.1102595 http://www.jimmunol.org/content/188/6/2905 Spatiotemporal Inhibition of Innate Immunity Signaling by the Tbc1d23 RAB-GAP Lesly De Arras, Ivana V. Yang, Brad Lackford, David W. H. Riches, Rytis Prekeris, Jonathan H. Freedman, David A. Schwartz and Scott Alper J Immunol cites 58 articles Submit online. Every submission reviewed by practicing scientists ? is published twice each month by Submit copyright permission requests at: http://www.aai.org/About/Publications/JI/copyright.html Receive free email-alerts when new articles cite this article. Sign up at: http://jimmunol.org/alerts http://jimmunol.org/subscription http://www.jimmunol.org/content/suppl/2012/02/06/jimmunol.110259 5.DC1 This article http://www.jimmunol.org/content/188/6/2905.full#ref-list-1 Information about subscribing to The JI No Triage! Fast Publication! Rapid Reviews! 30 days* Why • • • Material References Permissions Email Alerts Subscription Supplementary The Journal of Immunology The American Association of Immunologists, Inc., 1451 Rockville Pike, Suite 650, Rockville, MD 20852 All rights reserved. Print ISSN: 0022-1767 Online ISSN: 1550-6606. This information is current as of September 27, 2021. The Journal of Immunology Spatiotemporal Inhibition of Innate Immunity Signaling by the Tbc1d23 RAB-GAP Lesly De Arras,*,† Ivana V. Yang,†,‡ Brad Lackford,x David W. -

Retinoid-Induced Apoptosis in Normal and Neoplastic Tissues
Cell Death and Differentiation (1998) 5, 11 ± 19 1998 Stockton Press All rights reserved 13509047/98 $12.00 Review Retinoid-induced apoptosis in normal and neoplastic tissues Laszlo Nagy1,3,4, Vilmos A. Thomazy1, Richard A. Heyman2 retinoic acid receptor (RAR), which belongs to the superfamily and Peter J.A. Davies1,3 of ligand-activated transcription factors (nuclear receptors) revolutionized our understanding as to how retinoids exert 1 Department of Pharmacology, University of Texas-Houston, Medical School, their pleiotropic effects (for reviews see Chambon (1996); Houston, Texas 77225 USA Mangelsdorf et al (1994)). Members of the nuclear receptor 2 Ligand Pharmaceuticals, San Diego, California, 92121 USA superfamily mediate the biological effects of many hormones, 3 Corresponding author: PJAD, tel: 713-500-7480; fax: 713-500-7455; vitamins and drugs (i.e. steroid hormones, thyroid hormones, e-mail: [email protected] 4 vitamin D, prostaglandin-J (PG-J ) and drugs that activate Present address for correspondence: The Salk Institute for Biological Studies, 2 2 Gene Expression Laboratory, La Jolla, California 92037; peroxisomal proliferation). There are two families of retinoid tel: (619) 453-4100 fax:(619) 455-1349; e-mail: [email protected] receptors, Retinoid X Receptors (RXRs) that bind 9-cis retinoic acid (9-cis RA) and Retinoic Acid Receptors (RARs) Received 18.8.97; revised 19.9.97; accepted 22.9.97 that bind both 9-cis RA and all-trans retinoic acid (ATRA) (for Edited by M. Piacentini reviews see Chambon 1996; Mangelsdorf et al, 1994)). Each of these receptor families includes at least three distinct genes, (RARa,b and g; RXRa,b and g) that through differential Abstract promoter usage and alternative splicing, give rise to a large number of distinct retinoid receptor proteins (for reviews see Vitamin A and its derivatives (collectively referred to as Chambon 1996; Mangelsdorf et al, 1994). -

Retinoid X Receptor Is Necessary to Establish the S-Opsin Gradient In
Retinoid X Receptor ␥ Is Necessary to Establish the S-opsin Gradient in Cone Photoreceptors of the Developing Mouse Retina Melanie R. Roberts,1,2 Anita Hendrickson,2 Christopher R. McGuire,2 and Thomas A. Reh2 PURPOSE. The retinoid X receptors (RXRs) are members of the tion, they also have demonstrated some isoform-specific family of ligand-dependent nuclear hormone receptors. One of functions. For example, RXR␣ heterodimerizes with retinoic these genes, RXR␥, is expressed in highly restricted regions of acid receptors (RARs) to regulate retinal growth and cardiac the developing central nervous system (CNS), including the development.4 retina. Although previous studies have localized RXR␥ to de- Although all three isoforms are expressed in the developing veloping cone photoreceptors in several species, its function nervous system, RXR␥ has the most restricted and develop- in these cells is unknown. A prior study showed that thyroid mentally regulated expression. It is expressed in the develop- hormone receptor 2 (TR2) is necessary to establish proper ing striatum, part of the tegmentum, the pituitary, the ventral cone patterning in mice by activating medium-wavelength (M) horns of the spinal cord,3,5 and the retina.6 RXR␥-null mice cone opsin and suppressing short-wavelength (S) cone opsin. show isoform-specific defects in both striatal and hippocampal Thyroid hormone receptors often regulate gene transcription function, although RXR can partially compensate for the loss as heterodimeric complexes with RXRs. of RXR␥ in the striatum.2,7,8 RXR␥ has also been identified in 6,9,10 METHODS. To determine whether RXR␥ cooperates with TR2 the developing retina of Xenopus, chicks, and mice.