Identification of Regulatory RNA Elements Based on Structural
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

Functional Annotation of Exon Skipping Event in Human Pora Kim1,*,†, Mengyuan Yang1,†,Keyiya2, Weiling Zhao1 and Xiaobo Zhou1,3,4,*
D896–D907 Nucleic Acids Research, 2020, Vol. 48, Database issue Published online 23 October 2019 doi: 10.1093/nar/gkz917 ExonSkipDB: functional annotation of exon skipping event in human Pora Kim1,*,†, Mengyuan Yang1,†,KeYiya2, Weiling Zhao1 and Xiaobo Zhou1,3,4,* 1School of Biomedical Informatics, The University of Texas Health Science Center at Houston, Houston, TX 77030, USA, 2College of Electronics and Information Engineering, Tongji University, Shanghai, China, 3McGovern Medical School, The University of Texas Health Science Center at Houston, Houston, TX 77030, USA and 4School of Dentistry, The University of Texas Health Science Center at Houston, Houston, TX 77030, USA Received August 13, 2019; Revised September 21, 2019; Editorial Decision October 03, 2019; Accepted October 03, 2019 ABSTRACT been used as therapeutic targets (3–8). For example, MET has lost the binding site of E3 ubiquitin ligase CBL through Exon skipping (ES) is reported to be the most com- exon 14 skipping event (9), resulting in an enhanced expres- mon alternative splicing event due to loss of func- sion level of MET. MET amplification drives the prolifera- tional domains/sites or shifting of the open read- tion of tumor cells. Multiple tyrosine kinase inhibitors, such ing frame (ORF), leading to a variety of human dis- as crizotinib, cabozantinib and capmatinib, have been used eases and considered therapeutic targets. To date, to treat patients with MET exon 14 skipping (10). Another systematic and intensive annotations of ES events example is the dystrophin gene (DMD) in Duchenne mus- based on the skipped exon units in cancer and cular dystrophy (DMD), a progressive neuromuscular dis- normal tissues are not available. -

Genome-Wide Approach to Identify Risk Factors for Therapy-Related Myeloid Leukemia
Leukemia (2006) 20, 239–246 & 2006 Nature Publishing Group All rights reserved 0887-6924/06 $30.00 www.nature.com/leu ORIGINAL ARTICLE Genome-wide approach to identify risk factors for therapy-related myeloid leukemia A Bogni1, C Cheng2, W Liu2, W Yang1, J Pfeffer1, S Mukatira3, D French1, JR Downing4, C-H Pui4,5,6 and MV Relling1,6 1Department of Pharmaceutical Sciences, The University of Tennessee, Memphis, TN, USA; 2Department of Biostatistics, The University of Tennessee, Memphis, TN, USA; 3Hartwell Center, The University of Tennessee, Memphis, TN, USA; 4Department of Pathology, The University of Tennessee, Memphis, TN, USA; 5Department of Hematology/Oncology St Jude Children’s Research Hospital, The University of Tennessee, Memphis, TN, USA; and 6Colleges of Medicine and Pharmacy, The University of Tennessee, Memphis, TN, USA Using a target gene approach, only a few host genetic risk therapy increases, the importance of identifying host factors for factors for treatment-related myeloid leukemia (t-ML) have been secondary neoplasms increases. defined. Gene expression microarrays allow for a more 4 genome-wide approach to assess possible genetic risk factors Because DNA microarrays interrogate multiple ( 10 000) for t-ML. We assessed gene expression profiles (n ¼ 12 625 genes in one experiment, they allow for a ‘genome-wide’ probe sets) in diagnostic acute lymphoblastic leukemic cells assessment of genes that may predispose to leukemogenesis. from 228 children treated on protocols that included leukemo- DNA microarray analysis of gene expression has been used to genic agents such as etoposide, 13 of whom developed t-ML. identify distinct expression profiles that are characteristic of Expression of 68 probes, corresponding to 63 genes, was different leukemia subtypes.13,14 Studies using this method have significantly related to risk of t-ML. -

Molecular Basis for the Distinct Cellular Functions of the Lsm1-7 and Lsm2-8 Complexes
bioRxiv preprint doi: https://doi.org/10.1101/2020.04.22.055376; this version posted April 23, 2020. The copyright holder for this preprint (which was not certified by peer review) is the author/funder, who has granted bioRxiv a license to display the preprint in perpetuity. It is made available under aCC-BY-NC-ND 4.0 International license. Molecular basis for the distinct cellular functions of the Lsm1-7 and Lsm2-8 complexes Eric J. Montemayor1,2, Johanna M. Virta1, Samuel M. Hayes1, Yuichiro Nomura1, David A. Brow2, Samuel E. Butcher1 1Department of Biochemistry, University of Wisconsin-Madison, Madison, WI, USA. 2Department of Biomolecular Chemistry, University of Wisconsin School of Medicine and Public Health, Madison, WI, USA. Correspondence should be addressed to E.J.M. ([email protected]) and S.E.B. ([email protected]). Abstract Eukaryotes possess eight highly conserved Lsm (like Sm) proteins that assemble into circular, heteroheptameric complexes, bind RNA, and direct a diverse range of biological processes. Among the many essential functions of Lsm proteins, the cytoplasmic Lsm1-7 complex initiates mRNA decay, while the nuclear Lsm2-8 complex acts as a chaperone for U6 spliceosomal RNA. It has been unclear how these complexes perform their distinct functions while differing by only one out of seven subunits. Here, we elucidate the molecular basis for Lsm-RNA recognition and present four high-resolution structures of Lsm complexes bound to RNAs. The structures of Lsm2-8 bound to RNA identify the unique 2′,3′ cyclic phosphate end of U6 as a prime determinant of specificity. In contrast, the Lsm1-7 complex strongly discriminates against cyclic phosphates and tightly binds to oligouridylate tracts with terminal purines. -

MAP4K4 Expression in Cardiomyocytes: Multiple Isoforms, Multiple Phosphorylations and Interactions with Striatins
MAP4K4 expression in cardiomyocytes: multiple isoforms, multiple phosphorylations and interactions with striatins Article Published Version Creative Commons: Attribution 4.0 (CC-BY) Open access Fuller, S. J., Edmunds, N. S., McGuffin, L. J. ORCID: https://orcid.org/0000-0003-4501-4767, Hardyman, M. A., Cull, J. J., Alharbi, H. O., Meijles, D. N., Sugden, P. H. and Clerk, A. ORCID: https://orcid.org/0000-0002-5658-0708 (2021) MAP4K4 expression in cardiomyocytes: multiple isoforms, multiple phosphorylations and interactions with striatins. Biochemical Journal, 478 (11). pp. 2121-2143. ISSN 0264- 6021 doi: https://doi.org/10.1042/BCJ20210003 Available at http://centaur.reading.ac.uk/98442/ It is advisable to refer to the publisher’s version if you intend to cite from the work. See Guidance on citing . Published version at: http://dx.doi.org/10.1042/BCJ20210003 To link to this article DOI: http://dx.doi.org/10.1042/BCJ20210003 Publisher: Portland Press All outputs in CentAUR are protected by Intellectual Property Rights law, including copyright law. Copyright and IPR is retained by the creators or other copyright holders. Terms and conditions for use of this material are defined in the End User Agreement . www.reading.ac.uk/centaur CentAUR Central Archive at the University of Reading Reading’s research outputs online Biochemical Journal (2021) 478 2121–2143 https://doi.org/10.1042/BCJ20210003 Research Article MAP4K4 expression in cardiomyocytes: multiple isoforms, multiple phosphorylations and interactions with striatins Stephen J. Fuller1, Nick S. Edmunds1, Liam J. McGuffin1, Michelle A. Hardyman1, Joshua J. Cull1, Hajed O. Alharbi1, Daniel N. Meijles2, Peter H. -
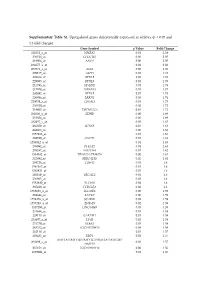
Supplementary Table S1. Upregulated Genes Differentially
Supplementary Table S1. Upregulated genes differentially expressed in athletes (p < 0.05 and 1.3-fold change) Gene Symbol p Value Fold Change 221051_s_at NMRK2 0.01 2.38 236518_at CCDC183 0.00 2.05 218804_at ANO1 0.00 2.05 234675_x_at 0.01 2.02 207076_s_at ASS1 0.00 1.85 209135_at ASPH 0.02 1.81 228434_at BTNL9 0.03 1.81 229985_at BTNL9 0.01 1.79 215795_at MYH7B 0.01 1.78 217979_at TSPAN13 0.01 1.77 230992_at BTNL9 0.01 1.75 226884_at LRRN1 0.03 1.74 220039_s_at CDKAL1 0.01 1.73 236520_at 0.02 1.72 219895_at TMEM255A 0.04 1.72 201030_x_at LDHB 0.00 1.69 233824_at 0.00 1.69 232257_s_at 0.05 1.67 236359_at SCN4B 0.04 1.64 242868_at 0.00 1.63 1557286_at 0.01 1.63 202780_at OXCT1 0.01 1.63 1556542_a_at 0.04 1.63 209992_at PFKFB2 0.04 1.63 205247_at NOTCH4 0.01 1.62 1554182_at TRIM73///TRIM74 0.00 1.61 232892_at MIR1-1HG 0.02 1.61 204726_at CDH13 0.01 1.6 1561167_at 0.01 1.6 1565821_at 0.01 1.6 210169_at SEC14L5 0.01 1.6 236963_at 0.02 1.6 1552880_at SEC16B 0.02 1.6 235228_at CCDC85A 0.02 1.6 1568623_a_at SLC35E4 0.00 1.59 204844_at ENPEP 0.00 1.59 1552256_a_at SCARB1 0.02 1.59 1557283_a_at ZNF519 0.02 1.59 1557293_at LINC00969 0.03 1.59 231644_at 0.01 1.58 228115_at GAREM1 0.01 1.58 223687_s_at LY6K 0.02 1.58 231779_at IRAK2 0.03 1.58 243332_at LOC105379610 0.04 1.58 232118_at 0.01 1.57 203423_at RBP1 0.02 1.57 AMY1A///AMY1B///AMY1C///AMY2A///AMY2B// 208498_s_at 0.03 1.57 /AMYP1 237154_at LOC101930114 0.00 1.56 1559691_at 0.01 1.56 243481_at RHOJ 0.03 1.56 238834_at MYLK3 0.01 1.55 213438_at NFASC 0.02 1.55 242290_at TACC1 0.04 1.55 ANKRD20A1///ANKRD20A12P///ANKRD20A2/// -

Bppart and Bpmax: RNA-RNA Interaction Partition Function and Structure Prediction for the Base Pair Counting Model
BPPart and BPMax: RNA-RNA Interaction Partition Function and Structure Prediction for the Base Pair Counting Model Ali Ebrahimpour-Boroojeny, Sanjay Rajopadhye, and Hamidreza Chitsaz ∗ Department of Computer Science, Colorado State University Abstract A few elite classes of RNA-RNA interaction (RRI), with complex roles in cellular functions such as miRNA-target and lncRNAs in human health, have already been studied. Accordingly, RRI bioinfor- matics tools tailored for those elite classes have been proposed in the last decade. Interestingly, there are somewhat unnoticed mRNA-mRNA interactions in the literature with potentially drastic biological roles. Hence, there is a need for high-throughput generic RRI bioinformatics tools. We revisit our RRI partition function algorithm, piRNA, which happens to be the most comprehensive and computationally-intensive thermodynamic model for RRI. We propose simpler models that are shown to retain the vast majority of the thermodynamic information that piRNA captures. We simplify the energy model and instead consider only weighted base pair counting to obtain BPPart for Base-pair Partition function and BPMax for Base-pair Maximization which are 225 and 1350 faster ◦ × × than piRNA, with a correlation of 0.855 and 0.836 with piRNA at 37 C on 50,500 experimentally charac- terized RRIs. This correlation increases to 0.920 and 0.904, respectively, at 180◦C. − Finally, we apply our algorithm BPPart to discover two disease-related RNAs, SNORD3D and TRAF3, and hypothesize their potential roles in Parkinson's disease and Cerebral Autosomal Dominant Arteri- opathy with Subcortical Infarcts and Leukoencephalopathy (CADASIL). 1 Introduction Since mid 1990s with the advent of RNA interference discovery, RNA-RNA interaction (RRI) has moved to the spotlight in modern, post-genome biology. -

Human Regulatory T Cells Mediate Transcriptional Modulation of Dendritic Cell Function
Human Regulatory T Cells Mediate Transcriptional Modulation of Dendritic Cell Function This information is current as Emily Mavin, Lindsay Nicholson, Syed Rafez Ahmed, Fei of September 24, 2021. Gao, Anne Dickinson and Xiao-nong Wang J Immunol published online 28 November 2016 http://www.jimmunol.org/content/early/2016/11/26/jimmun ol.1502487 Downloaded from Supplementary http://www.jimmunol.org/content/suppl/2016/11/26/jimmunol.150248 Material 7.DCSupplemental http://www.jimmunol.org/ Why The JI? Submit online. • Rapid Reviews! 30 days* from submission to initial decision • No Triage! Every submission reviewed by practicing scientists • Fast Publication! 4 weeks from acceptance to publication by guest on September 24, 2021 *average Subscription Information about subscribing to The Journal of Immunology is online at: http://jimmunol.org/subscription Permissions Submit copyright permission requests at: http://www.aai.org/About/Publications/JI/copyright.html Email Alerts Receive free email-alerts when new articles cite this article. Sign up at: http://jimmunol.org/alerts The Journal of Immunology is published twice each month by The American Association of Immunologists, Inc., 1451 Rockville Pike, Suite 650, Rockville, MD 20852 Copyright © 2016 by The American Association of Immunologists, Inc. All rights reserved. Print ISSN: 0022-1767 Online ISSN: 1550-6606. Published November 28, 2016, doi:10.4049/jimmunol.1502487 The Journal of Immunology Human Regulatory T Cells Mediate Transcriptional Modulation of Dendritic Cell Function Emily Mavin,*,1 Lindsay Nicholson,*,1 Syed Rafez Ahmed,* Fei Gao,† Anne Dickinson,* and Xiao-nong Wang* Regulatory T cells (Treg) attenuate dendritic cell (DC) maturation and stimulatory function. Current knowledge on the functional impact of semimature DC is limited to CD4+ T cell proliferation and cytokine production. -

Single-Cell RNA Sequencing Identifies Senescent Cerebromicrovascular Endothelial Cells in the Aged Mouse Brain
GeroScience (2020) 42:429–444 https://doi.org/10.1007/s11357-020-00177-1 ORIGINAL ARTICLE/UNDERSTANDING SENESCENCE IN BRAIN AGING AND ALZHEIMER’SDISEASE Single-cell RNA sequencing identifies senescent cerebromicrovascular endothelial cells in the aged mouse brain Tamas Kiss & Ádám Nyúl-Tóth & Priya Balasubramanian & Stefano Tarantini & Chetan Ahire & Jordan DelFavero & Andriy Yabluchanskiy & Tamas Csipo & Eszter Farkas & Graham Wiley & Lori Garman & Anna Csiszar & Zoltan Ungvari Received: 31 January 2020 /Accepted: 1 March 2020 /Published online: 31 March 2020 # American Aging Association 2020 Abstract Age-related phenotypic changes of therapeutic exploitation of senolytic drugs in preclinical cerebromicrovascular endothelial cells lead to dysregula- studies. However, difficulties with the detection of senes- tion of cerebral blood flow and blood-brain barrier dis- cent endothelial cells in wild type mouse models of aging ruption, promoting the pathogenesis of vascular cognitive hinder the assessment of the efficiency of senolytic treat- impairment (VCI). In recent years, endothelial cell senes- ments. To detect senescent endothelial cells in the aging cence has emerged as a potential mechanism contributing mouse brain, we analyzed 4233 cells in fractions enriched to microvascular pathologies opening the avenue to the for cerebromicrovascular endothelial cells and other cells Tamas Kiss, Ádám Nyúl-Tóth, Priya Balasubramanian and Stefano Tarantini contributed equally to this work. T. Kiss : Á. Nyúl-Tóth : P. Balasubramanian : S. Tarantini : S. Tarantini : A. Yabluchanskiy : Z. Ungvari C. Ahire : J. DelFavero : A. Yabluchanskiy : T. Csipo : Department of Public Health, International Training Program in A. Csiszar (*) : Z. Ungvari Geroscience, Doctoral School of Basic and Translational Medicine, Department of Biochemistry and Molecular Biology, Reynolds Semmelweis University, Budapest, Hungary Oklahoma Center on Aging/Center for Geroscience and Healthy Brain Aging, Vascular Cognitive Impairment and T. -

Unbiased Identification of Trans Regulators of ADAR and A-To-I RNA Editing 2 3 Emily C
bioRxiv preprint doi: https://doi.org/10.1101/631200; this version posted May 8, 2019. The copyright holder for this preprint (which was not certified by peer review) is the author/funder, who has granted bioRxiv a license to display the preprint in perpetuity. It is made available under aCC-BY-NC-ND 4.0 International license. 1 Unbiased identification of trans regulators of ADAR and A-to-I RNA editing 2 3 Emily C. Freund1, Anne L. Sapiro1, Qin Li1, Sandra Linder1, James J. Moresco2, John R. Yates 4 III2, Jin Billy Li1 5 6 1 Department of Genetics, Stanford University, Stanford, CA, USA 7 2 Department of Molecular Medicine, 10550 North Torrey Pines Road, SR302, The Scripps 8 Research Institute, La Jolla, California 92037 9 10 Correspondence: [email protected] 11 12 13 Abstract 14 Adenosine-to-Inosine RNA editing is catalyzed by ADAR enzymes that deaminate adenosine to 15 inosine. While many RNA editing sites are known, few trans regulators have been identified. We 16 perform BioID followed by mass-spectrometry to identify trans regulators of ADAR1 and ADAR2 17 in HeLa and M17 neuroblastoma cells. We identify known and novel ADAR-interacting proteins. 18 Using ENCODE data we validate and characterize a subset of the novel interactors as global or 19 site-specific RNA editing regulators. Our set of novel trans regulators includes all four members 20 of the DZF-domain-containing family of proteins: ILF3, ILF2, STRBP, and ZFR. We show that 21 these proteins interact with each ADAR and modulate RNA editing levels. -

Neddylation: a Novel Modulator of the Tumor Microenvironment Lisha Zhou1,2*†, Yanyu Jiang3†, Qin Luo1, Lihui Li1 and Lijun Jia1*
Zhou et al. Molecular Cancer (2019) 18:77 https://doi.org/10.1186/s12943-019-0979-1 REVIEW Open Access Neddylation: a novel modulator of the tumor microenvironment Lisha Zhou1,2*†, Yanyu Jiang3†, Qin Luo1, Lihui Li1 and Lijun Jia1* Abstract Neddylation, a post-translational modification that adds an ubiquitin-like protein NEDD8 to substrate proteins, modulates many important biological processes, including tumorigenesis. The process of protein neddylation is overactivated in multiple human cancers, providing a sound rationale for its targeting as an attractive anticancer therapeutic strategy, as evidence by the development of NEDD8-activating enzyme (NAE) inhibitor MLN4924 (also known as pevonedistat). Neddylation inhibition by MLN4924 exerts significantly anticancer effects mainly by triggering cell apoptosis, senescence and autophagy. Recently, intensive evidences reveal that inhibition of neddylation pathway, in addition to acting on tumor cells, also influences the functions of multiple important components of the tumor microenvironment (TME), including immune cells, cancer-associated fibroblasts (CAFs), cancer-associated endothelial cells (CAEs) and some factors, all of which are crucial for tumorigenesis. Here, we briefly summarize the latest progresses in this field to clarify the roles of neddylation in the TME, thus highlighting the overall anticancer efficacy of neddylaton inhibition. Keywords: Neddylation, Tumor microenvironment, Tumor-derived factors, Cancer-associated fibroblasts, Cancer- associated endothelial cells, Immune cells Introduction Overall, binding of NEDD8 molecules to target proteins Neddylation is a reversible covalent conjugation of an can affect their stability, subcellular localization, conform- ubiquitin-like molecule NEDD8 (neuronal precursor ation and function [4]. The best-characterized substrates cell-expressed developmentally down-regulated protein of neddylation are the cullin subunits of Cullin-RING li- 8) to a lysine residue of the substrate protein [1, 2]. -

Characterization of a 7.6-Mb Germline Deletion Encompassing the NF1 Locus and About a Hundred Genes in an NF1 Contiguous Gene Syndrome Patient
European Journal of Human Genetics (2008) 16, 1459–1466 & 2008 Macmillan Publishers Limited All rights reserved 1018-4813/08 $32.00 www.nature.com/ejhg ARTICLE Characterization of a 7.6-Mb germline deletion encompassing the NF1 locus and about a hundred genes in an NF1 contiguous gene syndrome patient Eric Pasmant*,1,2, Aure´lie de Saint-Trivier2, Ingrid Laurendeau1, Anne Dieux-Coeslier3, Be´atrice Parfait1,2, Michel Vidaud1,2, Dominique Vidaud1,2 and Ivan Bie`che1,2 1UMR745 INSERM, Universite´ Paris Descartes, Faculte´ des Sciences Pharmaceutiques et Biologiques, Paris, France; 2Service de Biochimie et de Ge´ne´tique Mole´culaire, Hoˆpital Beaujon AP-HP, Clichy, France; 3Service de Ge´ne´tique Clinique, Hoˆpital Jeanne de Flandre, Lille, France We describe a large germline deletion removing the NF1 locus, identified by heterozygosity mapping based on microsatellite markers, in an 8-year-old French girl with a particularly severe NF1 contiguous gene syndrome. We used gene-dose mapping with sequence-tagged site real-time PCR to locate the deletion end points, which were precisely characterized by means of long-range PCR and nucleotide sequencing. The deletion is located on chromosome arm 17q and is exactly 7 586 986 bp long. It encompasses the entire NF1 locus and about 100 other genes, including numerous chemokine genes, an attractive in silico-selected cerebrally expressed candidate gene (designated NUFIP2, for nuclear fragile X mental retardation protein interacting protein 2; NM_020772) and four microRNA genes. Interestingly, the centromeric breakpoint is located in intron 4 of the PIPOX gene (pipecolic acid oxidase; NM_016518) and the telomeric breakpoint in intron 5 of the GGNBP2 gene (gametogenetin binding protein 2; NM_024835) coding a transcription factor. -

Noelia Díaz Blanco
Effects of environmental factors on the gonadal transcriptome of European sea bass (Dicentrarchus labrax), juvenile growth and sex ratios Noelia Díaz Blanco Ph.D. thesis 2014 Submitted in partial fulfillment of the requirements for the Ph.D. degree from the Universitat Pompeu Fabra (UPF). This work has been carried out at the Group of Biology of Reproduction (GBR), at the Department of Renewable Marine Resources of the Institute of Marine Sciences (ICM-CSIC). Thesis supervisor: Dr. Francesc Piferrer Professor d’Investigació Institut de Ciències del Mar (ICM-CSIC) i ii A mis padres A Xavi iii iv Acknowledgements This thesis has been made possible by the support of many people who in one way or another, many times unknowingly, gave me the strength to overcome this "long and winding road". First of all, I would like to thank my supervisor, Dr. Francesc Piferrer, for his patience, guidance and wise advice throughout all this Ph.D. experience. But above all, for the trust he placed on me almost seven years ago when he offered me the opportunity to be part of his team. Thanks also for teaching me how to question always everything, for sharing with me your enthusiasm for science and for giving me the opportunity of learning from you by participating in many projects, collaborations and scientific meetings. I am also thankful to my colleagues (former and present Group of Biology of Reproduction members) for your support and encouragement throughout this journey. To the “exGBRs”, thanks for helping me with my first steps into this world. Working as an undergrad with you Dr.