Insights Into the Biology of Candidate Division OP3 Lim Populations
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-
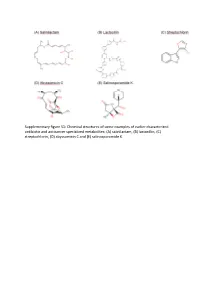
Chemical Structures of Some Examples of Earlier Characterized Antibiotic and Anticancer Specialized
Supplementary figure S1: Chemical structures of some examples of earlier characterized antibiotic and anticancer specialized metabolites: (A) salinilactam, (B) lactocillin, (C) streptochlorin, (D) abyssomicin C and (E) salinosporamide K. Figure S2. Heat map representing hierarchical classification of the SMGCs detected in all the metagenomes in the dataset. Table S1: The sampling locations of each of the sites in the dataset. Sample Sample Bio-project Site depth accession accession Samples Latitude Longitude Site description (m) number in SRA number in SRA AT0050m01B1-4C1 SRS598124 PRJNA193416 Atlantis II water column 50, 200, Water column AT0200m01C1-4D1 SRS598125 21°36'19.0" 38°12'09.0 700 and above the brine N "E (ATII 50, ATII 200, 1500 pool water layers AT0700m01C1-3D1 SRS598128 ATII 700, ATII 1500) AT1500m01B1-3C1 SRS598129 ATBRUCL SRS1029632 PRJNA193416 Atlantis II brine 21°36'19.0" 38°12'09.0 1996– Brine pool water ATBRLCL1-3 SRS1029579 (ATII UCL, ATII INF, N "E 2025 layers ATII LCL) ATBRINP SRS481323 PRJNA219363 ATIID-1a SRS1120041 PRJNA299097 ATIID-1b SRS1120130 ATIID-2 SRS1120133 2168 + Sea sediments Atlantis II - sediments 21°36'19.0" 38°12'09.0 ~3.5 core underlying ATII ATIID-3 SRS1120134 (ATII SDM) N "E length brine pool ATIID-4 SRS1120135 ATIID-5 SRS1120142 ATIID-6 SRS1120143 Discovery Deep brine DDBRINP SRS481325 PRJNA219363 21°17'11.0" 38°17'14.0 2026– Brine pool water N "E 2042 layers (DD INF, DD BR) DDBRINE DD-1 SRS1120158 PRJNA299097 DD-2 SRS1120203 DD-3 SRS1120205 Discovery Deep 2180 + Sea sediments sediments 21°17'11.0" -

The 2014 Golden Gate National Parks Bioblitz - Data Management and the Event Species List Achieving a Quality Dataset from a Large Scale Event
National Park Service U.S. Department of the Interior Natural Resource Stewardship and Science The 2014 Golden Gate National Parks BioBlitz - Data Management and the Event Species List Achieving a Quality Dataset from a Large Scale Event Natural Resource Report NPS/GOGA/NRR—2016/1147 ON THIS PAGE Photograph of BioBlitz participants conducting data entry into iNaturalist. Photograph courtesy of the National Park Service. ON THE COVER Photograph of BioBlitz participants collecting aquatic species data in the Presidio of San Francisco. Photograph courtesy of National Park Service. The 2014 Golden Gate National Parks BioBlitz - Data Management and the Event Species List Achieving a Quality Dataset from a Large Scale Event Natural Resource Report NPS/GOGA/NRR—2016/1147 Elizabeth Edson1, Michelle O’Herron1, Alison Forrestel2, Daniel George3 1Golden Gate Parks Conservancy Building 201 Fort Mason San Francisco, CA 94129 2National Park Service. Golden Gate National Recreation Area Fort Cronkhite, Bldg. 1061 Sausalito, CA 94965 3National Park Service. San Francisco Bay Area Network Inventory & Monitoring Program Manager Fort Cronkhite, Bldg. 1063 Sausalito, CA 94965 March 2016 U.S. Department of the Interior National Park Service Natural Resource Stewardship and Science Fort Collins, Colorado The National Park Service, Natural Resource Stewardship and Science office in Fort Collins, Colorado, publishes a range of reports that address natural resource topics. These reports are of interest and applicability to a broad audience in the National Park Service and others in natural resource management, including scientists, conservation and environmental constituencies, and the public. The Natural Resource Report Series is used to disseminate comprehensive information and analysis about natural resources and related topics concerning lands managed by the National Park Service. -

A Genomic View of Trophic and Metabolic Diversity in Clade-Specific Lamellodysidea Sponge Microbiomes
UC San Diego UC San Diego Previously Published Works Title A genomic view of trophic and metabolic diversity in clade-specific Lamellodysidea sponge microbiomes. Permalink https://escholarship.org/uc/item/6z2365ft Journal Microbiome, 8(1) ISSN 2049-2618 Authors Podell, Sheila Blanton, Jessica M Oliver, Aaron et al. Publication Date 2020-06-23 DOI 10.1186/s40168-020-00877-y Peer reviewed eScholarship.org Powered by the California Digital Library University of California Podell et al. Microbiome (2020) 8:97 https://doi.org/10.1186/s40168-020-00877-y RESEARCH Open Access A genomic view of trophic and metabolic diversity in clade-specific Lamellodysidea sponge microbiomes Sheila Podell1 , Jessica M. Blanton1, Aaron Oliver1, Michelle A. Schorn2, Vinayak Agarwal3, Jason S. Biggs4, Bradley S. Moore5,6,7 and Eric E. Allen1,5,7,8* Abstract Background: Marine sponges and their microbiomes contribute significantly to carbon and nutrient cycling in global reefs, processing and remineralizing dissolved and particulate organic matter. Lamellodysidea herbacea sponges obtain additional energy from abundant photosynthetic Hormoscilla cyanobacterial symbionts, which also produce polybrominated diphenyl ethers (PBDEs) chemically similar to anthropogenic pollutants of environmental concern. Potential contributions of non-Hormoscilla bacteria to Lamellodysidea microbiome metabolism and the synthesis and degradation of additional secondary metabolites are currently unknown. Results: This study has determined relative abundance, taxonomic novelty, metabolic -

Methanosaeta Pelagica" Sp
Aceticlastic and NaCl-Requiring Methanogen "Methanosaeta pelagica" sp. nov., Isolated from Marine Tidal Flat Title Sediment Author(s) Mori, Koji; Iino, Takao; Suzuki, Ken-Ichiro; Yamaguchi, Kaoru; Kamagata, Yoichi Applied and Environmental Microbiology, 78(9), 3416-3423 Citation https://doi.org/10.1128/AEM.07484-11 Issue Date 2012-05 Doc URL http://hdl.handle.net/2115/50384 Rights © 2012 by the American Society for Microbiology Type article (author version) File Information AEM78-9_3416-3423.pdf Instructions for use Hokkaido University Collection of Scholarly and Academic Papers : HUSCAP Mori et al. Title 2 Aceticlastic and NaCl-requiring methanogen “Methanosaeta pelagica” sp. nov., isolated from marine tidal flat sediment. 4 Authors 6 Koji Mori,1 Takao Iino,2 Ken-ichiro Suzuki,1 Kaoru Yamaguchi1 & Yoichi Kamagata3 1NITE Biological Resource Center (NBRC), National Institute of Technology and Evaluation (NITE), 8 2-5-8 Kazusakamatari, Kisarazu, Chiba 292-0818, Japan 2Japan Collection of Microorganisms, RIKEN BioResource Center, 2-1 Hirosawa, Wako, Saitama 10 351-0198, Japan 3Bioproduction Research Institute, National Institute of Advanced Industrial Science and Technology 12 (AIST), Central 6, 1-1-1 Higashi, Tsukuba, Ibaraki 400-8511, Japan 14 Corresponding author Koji Mori 16 NITE Biological Resource Center (NBRC), National Institute of Technology and Evaluation (NITE), 2-5-8 Kazusakamatari, Kisarazu, Chiba 292-0818, Japan 18 Tel, +81-438-20-5763; Fax, +81-438-52-2329; E-mail, [email protected] 20 Running title Marine aceticlastic methanogen, Methanosaeta pelagica 22 1 Mori et al. 2 ABSTRACT Acetate is a key compound for anaerobic organic matter degradation, and so far, two genera, 4 Methanosaeta and Methanosarcina, are only contributors for acetate degradation among methanogens. -

Alpine Soil Bacterial Community and Environmental Filters Bahar Shahnavaz
Alpine soil bacterial community and environmental filters Bahar Shahnavaz To cite this version: Bahar Shahnavaz. Alpine soil bacterial community and environmental filters. Other [q-bio.OT]. Université Joseph-Fourier - Grenoble I, 2009. English. tel-00515414 HAL Id: tel-00515414 https://tel.archives-ouvertes.fr/tel-00515414 Submitted on 6 Sep 2010 HAL is a multi-disciplinary open access L’archive ouverte pluridisciplinaire HAL, est archive for the deposit and dissemination of sci- destinée au dépôt et à la diffusion de documents entific research documents, whether they are pub- scientifiques de niveau recherche, publiés ou non, lished or not. The documents may come from émanant des établissements d’enseignement et de teaching and research institutions in France or recherche français ou étrangers, des laboratoires abroad, or from public or private research centers. publics ou privés. THÈSE Pour l’obtention du titre de l'Université Joseph-Fourier - Grenoble 1 École Doctorale : Chimie et Sciences du Vivant Spécialité : Biodiversité, Écologie, Environnement Communautés bactériennes de sols alpins et filtres environnementaux Par Bahar SHAHNAVAZ Soutenue devant jury le 25 Septembre 2009 Composition du jury Dr. Thierry HEULIN Rapporteur Dr. Christian JEANTHON Rapporteur Dr. Sylvie NAZARET Examinateur Dr. Jean MARTIN Examinateur Dr. Yves JOUANNEAU Président du jury Dr. Roberto GEREMIA Directeur de thèse Thèse préparée au sien du Laboratoire d’Ecologie Alpine (LECA, UMR UJF- CNRS 5553) THÈSE Pour l’obtention du titre de Docteur de l’Université de Grenoble École Doctorale : Chimie et Sciences du Vivant Spécialité : Biodiversité, Écologie, Environnement Communautés bactériennes de sols alpins et filtres environnementaux Bahar SHAHNAVAZ Directeur : Roberto GEREMIA Soutenue devant jury le 25 Septembre 2009 Composition du jury Dr. -

Bradymonabacteria, a Novel Bacterial Predator Group with Versatile
Mu et al. Microbiome (2020) 8:126 https://doi.org/10.1186/s40168-020-00902-0 RESEARCH Open Access Bradymonabacteria, a novel bacterial predator group with versatile survival strategies in saline environments Da-Shuai Mu1,2, Shuo Wang2, Qi-Yun Liang2, Zhao-Zhong Du2, Renmao Tian3, Yang Ouyang3, Xin-Peng Wang2, Aifen Zhou3, Ya Gong1,2, Guan-Jun Chen1,2, Joy Van Nostrand3, Yunfeng Yang4, Jizhong Zhou3,4 and Zong-Jun Du1,2* Abstract Background: Bacterial predation is an important selective force in microbial community structure and dynamics. However, only a limited number of predatory bacteria have been reported, and their predatory strategies and evolutionary adaptations remain elusive. We recently isolated a novel group of bacterial predators, Bradymonabacteria, representative of the novel order Bradymonadales in δ-Proteobacteria. Compared with those of other bacterial predators (e.g., Myxococcales and Bdellovibrionales), the predatory and living strategies of Bradymonadales are still largely unknown. Results: Based on individual coculture of Bradymonabacteria with 281 prey bacteria, Bradymonabacteria preyed on diverse bacteria but had a high preference for Bacteroidetes. Genomic analysis of 13 recently sequenced Bradymonabacteria indicated that these bacteria had conspicuous metabolic deficiencies, but they could synthesize many polymers, such as polyphosphate and polyhydroxyalkanoates. Dual transcriptome analysis of cocultures of Bradymonabacteria and prey suggested a potential contact-dependent predation mechanism. Comparative genomic analysis with 24 other bacterial predators indicated that Bradymonabacteria had different predatory and living strategies. Furthermore, we identified Bradymonadales from 1552 publicly available 16S rRNA amplicon sequencing samples, indicating that Bradymonadales was widely distributed and highly abundant in saline environments. Phylogenetic analysis showed that there may be six subgroups in this order; each subgroup occupied a different habitat. -

Single-Cell Genomics Reveals the Lifestyle of Poribacteria, a Candidate Phylum Symbiotically Associated with Marine Sponges
The ISME Journal (2011) 5, 61–70 & 2011 International Society for Microbial Ecology All rights reserved 1751-7362/11 www.nature.com/ismej ORIGINAL ARTICLE Single-cell genomics reveals the lifestyle of Poribacteria, a candidate phylum symbiotically associated with marine sponges Alexander Siegl1,5, Janine Kamke1, Thomas Hochmuth2,Jo¨rn Piel2, Michael Richter3, Chunguang Liang4, Thomas Dandekar4 and Ute Hentschel1 1Department of Botany II, Julius-von-Sachs Institute for Biological Sciences, University of Wuerzburg, Wuerzburg, Germany; 2Kekule´ Institute of Organic Chemistry and Biochemistry, University of Bonn, Bonn, Germany; 3Ribocon GmbH, Bremen, Germany and 4Department of Bioinformatics, Biocenter, University of Wuerzburg, Wuerzburg, Germany In this study, we present a single-cell genomics approach for the functional characterization of the candidate phylum Poribacteria, members of which are nearly exclusively found in marine sponges. The microbial consortia of the Mediterranean sponge Aplysina aerophoba were singularized by fluorescence-activated cell sorting, and individual microbial cells were subjected to phi29 polymerase-mediated ‘whole-genome amplification’. Pyrosequencing of a single amplified genome (SAG) derived from a member of the Poribacteria resulted in nearly 1.6 Mb of genomic information distributed among 554 contigs analyzed in this study. Approximately two-third of the poribacterial genome was sequenced. Our findings shed light on the functional properties and lifestyle of a possibly ancient bacterial symbiont of marine sponges. The Poribacteria are mixotrophic bacteria with autotrophic CO2-fixation capacities through the Wood–Ljungdahl pathway. The cell wall is of Gram-negative origin. The Poribacteria produce at least two polyketide synthases (PKSs), one of which is the sponge-specific Sup-type PKS. -

New 16S Rrna Primers to Uncover Bdellovibrio and Like Organisms Diversity and Abundance Jade Ezzedine, Cécile Chardon, Stéphan Jacquet
New 16S rRNA primers to uncover Bdellovibrio and like organisms diversity and abundance Jade Ezzedine, Cécile Chardon, Stéphan Jacquet To cite this version: Jade Ezzedine, Cécile Chardon, Stéphan Jacquet. New 16S rRNA primers to uncover Bdellovibrio and like organisms diversity and abundance. Journal of Microbiological Methods, Elsevier, 2020, 10.1016/j.mimet.2020.105996. hal-02935301 HAL Id: hal-02935301 https://hal.inrae.fr/hal-02935301 Submitted on 10 Sep 2020 HAL is a multi-disciplinary open access L’archive ouverte pluridisciplinaire HAL, est archive for the deposit and dissemination of sci- destinée au dépôt et à la diffusion de documents entific research documents, whether they are pub- scientifiques de niveau recherche, publiés ou non, lished or not. The documents may come from émanant des établissements d’enseignement et de teaching and research institutions in France or recherche français ou étrangers, des laboratoires abroad, or from public or private research centers. publics ou privés. Journal of Microbiological Methods 175 (2020) 105996 Contents lists available at ScienceDirect Journal of Microbiological Methods journal homepage: www.elsevier.com/locate/jmicmeth New 16S rRNA primers to uncover Bdellovibrio and like organisms diversity T and abundance ⁎ Jade A. Ezzedine, Cécile Chardon, Stéphan Jacquet Université Savoie Mont-Blanc, INRAE, UMR CARRTEL, Thonon-les-Bains, France ARTICLE INFO ABSTRACT Keywords: Appropriate use and specific primers are important in assessing the diversity and abundance of microbial groups Bdellovibrio and like organisms of interest. Bdellovibrio and like organisms (BALOs), that refer to obligate Gram-negative bacterial predators of Primer design other Gram-negative bacteria, evolved in terms of taxonomy and classification over the past two decades. -

Insights Into Archaeal Evolution and Symbiosis from the Genomes of a Nanoarchaeon and Its Inferred Crenarchaeal Host from Obsidian Pool, Yellowstone National Park
University of Tennessee, Knoxville TRACE: Tennessee Research and Creative Exchange Microbiology Publications and Other Works Microbiology 4-22-2013 Insights into archaeal evolution and symbiosis from the genomes of a nanoarchaeon and its inferred crenarchaeal host from Obsidian Pool, Yellowstone National Park Mircea Podar University of Tennessee - Knoxville, [email protected] Kira S. Makarova National Institutes of Health David E. Graham University of Tennessee - Knoxville, [email protected] Yuri I. Wolf National Institutes of Health Eugene V. Koonin National Institutes of Health See next page for additional authors Follow this and additional works at: https://trace.tennessee.edu/utk_micrpubs Part of the Microbiology Commons Recommended Citation Biology Direct 2013, 8:9 doi:10.1186/1745-6150-8-9 This Article is brought to you for free and open access by the Microbiology at TRACE: Tennessee Research and Creative Exchange. It has been accepted for inclusion in Microbiology Publications and Other Works by an authorized administrator of TRACE: Tennessee Research and Creative Exchange. For more information, please contact [email protected]. Authors Mircea Podar, Kira S. Makarova, David E. Graham, Yuri I. Wolf, Eugene V. Koonin, and Anna-Louise Reysenbach This article is available at TRACE: Tennessee Research and Creative Exchange: https://trace.tennessee.edu/ utk_micrpubs/44 Podar et al. Biology Direct 2013, 8:9 http://www.biology-direct.com/content/8/1/9 RESEARCH Open Access Insights into archaeal evolution and symbiosis from the genomes of a nanoarchaeon and its inferred crenarchaeal host from Obsidian Pool, Yellowstone National Park Mircea Podar1,2*, Kira S Makarova3, David E Graham1,2, Yuri I Wolf3, Eugene V Koonin3 and Anna-Louise Reysenbach4 Abstract Background: A single cultured marine organism, Nanoarchaeum equitans, represents the Nanoarchaeota branch of symbiotic Archaea, with a highly reduced genome and unusual features such as multiple split genes. -

Analyzing Metagenome Data Obtained by High-Throughput Sequencing
Centrum für Biotechnologie Analyzing Metagenome Data Obtained by High-Throughput Sequencing A. Pühler Center for Biotechnology Bielefeld University International Conference: Getting Post 2010 Biodiversity Targets Right Bragança Paulista/SP, Brazil December 11th – 15th, 2010 Content of Talk • Sequence analysis of the metagenome of a model microbial community • Analysis of assembled contigs and single reads by the help of completely sequenced genomes • The functional and taxonomic analysis of single reads using the software programs MetaSAMS and CARMA • The taxonomic analysis of a model microbial community based on 16S-rDNA sequences Sequence Analysis of the Metagenome of a Model Microbial Community (Part I) • Sequencing devices at the CeBiTec of Bielefeld University • Introduction of the model microbial community residing in an agrigultural biogas production • Sequence analysis of the metagenome of the model microbial community High-Throughput Sequencing Devices at the CeBiTec of Bielefeld University Sequencing techniques high-throughput sequencing ABI 3730xl DNA Genome Sequencer Genome Analyzer Analyzer (Applied GS FLX (Roche) (Illumina, Inc.) Biosystems) Genomics Platform Bioinformatics expertise and environment professional data evaluation Bioinformatics Platform Comparison of Different Sequencing Technologies Sequencing techniques ABI 3730xl DNA Genome Sequencer Genome Analyzer Analyzer (Applied GS FLX (Roche) (Illumina, Inc.) Biosystems) read length: 1100 bp 400 bp 150 bp sequenced bases/run: 0,1 Mb 500 Mb 45 Gb The GS FLX system is evidently best suited for a metagenome analysis since it offers long read length combined with an acceptable output. Metagenome Analysis of a Model Microbial Community Residing in a Biogas Production Plant Using Ultrafast Sequencing Biogas production from primary renewable products Biogas is produced during anaerobic digestion of biomass by specific microbial consortia Characteristics of the Analyzed Biogas Plant Located Close to the City of Bielefeld . -

An Integrated Study on Microbial Community in Anaerobic Digestion Systems
An Integrated Study on Microbial Community in Anaerobic Digestion Systems DISSERTATION Presented in Partial Fulfillment of the Requirements for the Degree Doctor of Philosophy in the Graduate School of The Ohio State University By Yueh-Fen Li Graduate Program in Environmental Science The Ohio State University 2013 Dissertation Committee: Dr. Zhongtang Yu, Advisor Dr. Brian Ahmer Dr. Richard Dick Dr. Olli Tuovinen Copyrighted by Yueh-Fen Li 2013 Abstract Anaerobic digestion (AD) is an attractive microbiological technology for both waste treatment and energy production. Microorganisms are the driving force for the whole transformation process in anaerobic digesters. However, the microbial community underpinning the AD process remains poorly understood, especially with respect to community composition and dynamics in response to variations in feedstocks and operations. The overall objective was to better understand the microbiology driving anaerobic digestion processes by systematically investigating the diversity, composition and succession of microbial communities, both bacterial and archaeal, in anaerobic digesters of different designs, fed different feedstocks, and operated under different conditions. The first two studies focused on propionate-degrading bacteria with an emphasis on syntrophic propionate-oxidizing bacteria. Propionate is one of the most important intermediates and has great influence on AD stability in AD systems because it is inhibitory to methanogens and it can only be metabolized through syntrophic propionate- oxidizing acetogenesis under methanogenic conditions. In the first study (chapter 3), primers specific to the propionate-CoA transferase gene (pct) were designed and used to construct clone libraries, which were sequenced and analyzed to investigate the diversity and distribution of propionate-utilizing bacteria present in the granular and the liquid portions of samples collected from four digesters of different designs, fed different ii feedstocks, and operated at different temperatures. -

Methanogens Diversity During Anaerobic Sewage Sludge Stabilization and the Effect of Temperature
processes Article Methanogens Diversity during Anaerobic Sewage Sludge Stabilization and the Effect of Temperature Tomáš Vítˇez 1,2, David Novák 3, Jan Lochman 3,* and Monika Vítˇezová 1,* 1 Department of Experimental Biology, Faculty of Science, Masaryk University, 62500 Brno, Czech Republic; [email protected] 2 Department of Agricultural, Food and Environmental Engineering, Faculty of AgriSciences, Mendel University, 61300 Brno, Czech Republic 3 Department of Biochemistry, Faculty of Science, Masaryk University, 62500 Brno, Czech Republic; [email protected] * Correspondence: [email protected] (J.L.); [email protected] (M.V.); Tel.: +420-549-495-602 (J.L.); Tel.: +420-549-497-177 (M.V.) Received: 29 June 2020; Accepted: 10 July 2020; Published: 12 July 2020 Abstract: Anaerobic sludge stabilization is a commonly used technology. Most fermenters are operated at a mesophilic temperature regime. Modern trends in waste management aim to minimize waste generation. One of the strategies can be achieved by anaerobically stabilizing the sludge by raising the temperature. Higher temperatures will allow faster decomposition of organic matter, shortening the retention time, and increasing biogas production. This work is focused on the description of changes in the community of methanogenic microorganisms at different temperatures during the sludge stabilization. At higher temperatures, biogas contained a higher percentage of methane, however, there was an undesirable accumulation of ammonia in the fermenter. Representatives of the hydrogenotrophic genus Methanoliea were described at all temperatures tested. At temperatures up to 50 ◦C, a significant proportion of methanogens were also formed by acetoclastic representatives of Methanosaeta sp. and acetoclastic representatives of the order Methanosarcinales.