Epigenetic Mechanism of CENP-A Loading to Centromeres Muhammad Shuaib
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

Datasheet for Histone H2B Human, Recombinant (M2505; Lot 0031404)
Supplied in: 20 mM Sodium Phosphate (pH 7.0), Quality Control Assays: Ionization-Time of Flight Mass Spectrometry). The Histone H2B 300 mM NaCl and 1 mM EDTA. SDS-PAGE: 0.5 µg, 1.0 µg, 2.0 µg, 5.0 µg, average mass calculated from primary sequence Human, Recombinant 10.0 µg Histone H2B Human, Recombinant were is 13788.97 Da. This confirms the protein identity Note: The protein concentration (1 mg/ml, 73 µM) loaded on a 10–20% Tris-Glycine SDS-PAGE gel as well as the absence of any modifications of the is calculated using the molar extinction coefficient and stained with Coomassie Blue. The calculated histone. 1-800-632-7799 for Histone H2B (6400) and its absorbance at molecular weight is 13788.97 Da. Its apparent [email protected] 280 nm (3,4). 1.0 A units = 2.2 mg/ml N-terminal Protein Sequencing: Protein identity 280 molecular weight on 10–20% Tris-Glycine SDS- www.neb.com was confirmed using Edman Degradation to PAGE gel is ~17 kDa. M2505S 003140416041 Synonyms: Histone H2B/q, Histone H2B.1, sequence the intact protein. Histone H2B-GL105 Mass Spectrometry: The mass of purified Histone H2B Human, Recombinant is 13788.5 Da Protease Assay: After incubation of 10 µg of M2505S B r kDa 1 2 3 4 5 6 7 as determined by ESI-TOF MS (Electrospray Histone H2B Human, Recombinant with a standard 250 4.0 mixture of proteins for 2 hours at 37°C, no 100 µg 1.0 mg/ml Lot: 0031404 150 13788.5 100 proteolytic activity could be detected by SDS- RECOMBINANT Store at –20°C Exp: 4/16 80 PAGE. -

The C-Terminal Region of Histone H4 Recognized by Antinucleosome Th Cells in Identification of a Minimal T Cell Epitope
Identification of a Minimal T Cell Epitope Recognized by Antinucleosome Th Cells in the C-Terminal Region of Histone H4 This information is current as Patrice Decker, Anne Le Moal, Jean-Paul Briand and of October 2, 2021. Sylviane Muller J Immunol 2000; 165:654-662; ; doi: 10.4049/jimmunol.165.2.654 http://www.jimmunol.org/content/165/2/654 Downloaded from References This article cites 34 articles, 13 of which you can access for free at: http://www.jimmunol.org/content/165/2/654.full#ref-list-1 http://www.jimmunol.org/ Why The JI? Submit online. • Rapid Reviews! 30 days* from submission to initial decision • No Triage! Every submission reviewed by practicing scientists • Fast Publication! 4 weeks from acceptance to publication *average by guest on October 2, 2021 Subscription Information about subscribing to The Journal of Immunology is online at: http://jimmunol.org/subscription Permissions Submit copyright permission requests at: http://www.aai.org/About/Publications/JI/copyright.html Email Alerts Receive free email-alerts when new articles cite this article. Sign up at: http://jimmunol.org/alerts The Journal of Immunology is published twice each month by The American Association of Immunologists, Inc., 1451 Rockville Pike, Suite 650, Rockville, MD 20852 Copyright © 2000 by The American Association of Immunologists All rights reserved. Print ISSN: 0022-1767 Online ISSN: 1550-6606. Identification of a Minimal T Cell Epitope Recognized by Antinucleosome Th Cells in the C-Terminal Region of Histone H41 Patrice Decker, Anne Le Moal, Jean-Paul Briand, and Sylviane Muller2 Autoreactive T cells responding to systemic autoantigens have been characterized in patients and mice with autoimmune diseases and in healthy individuals. -

Y-Chromosome Short Tandem Repeat, Typing Technology, Locus Information and Allele Frequency in Different Population: a Review
Vol. 14(27), pp. 2175-2178, 8 July, 2015 DOI:10.5897/AJB2015.14457 Article Number: 2E48C3F54052 ISSN 1684-5315 African Journal of Biotechnology Copyright © 2015 Author(s) retain the copyright of this article http://www.academicjournals.org/AJB Review Y-Chromosome short tandem repeat, typing technology, locus information and allele frequency in different population: A review Muhanned Abdulhasan Kareem1, Ameera Omran Hussein2 and Imad Hadi Hameed2* 1Babylon University, Centre of Environmental Research, Hilla City, Iraq. 2Department of Molecular Biology, Babylon University, Hilla City, Iraq. Received 29 January, 2015; Accepted 29 June, 2015 Chromosome Y microsatellites seem to be ideal markers to delineate differences between human populations. They are transmitted in uniparental and they are very sensitive for genetic drift. This review will highlight the importance of the Y- Chromosome as a tool for tracing human evolution and describes some details of Y-chromosomal short tandem repeat (STR) analysis. Among them are: microsatellites, amplification using polymerase chain reaction (PCR) of STRs, separation and detection and advantages of X-chromosomal microsatellites. Key words: Forensic, population, review, STR, Y- chromosome. INTRODUCTION Microsatellites are DNA regions with repeat units that are microsatellite, but repeats of five (penta-) or six (hexa-) 2 to 7 bp in length or most generally short tandem nucleotides are usually classified as microsatellites as repeats (STRs) or simple sequence repeats (SSRs) well. DNA can be used to study human evolution. (Ellegren, 2000; Imad et al., 2014). The classification of Besides, information from DNA typing is important for the DNA sequences is determined by the length of the medico-legal matters with polymorphisms leading to more core repeat unit and the number of adjacent repeat units. -

Histone Variants: Deviants?
Downloaded from genesdev.cshlp.org on September 25, 2021 - Published by Cold Spring Harbor Laboratory Press REVIEW Histone variants: deviants? Rohinton T. Kamakaka2,3 and Sue Biggins1 1Division of Basic Sciences, Fred Hutchinson Cancer Research Center, Seattle, Washington 98109, USA; 2UCT/National Institutes of Health, Bethesda, Maryland 20892, USA Histones are a major component of chromatin, the pro- sealing the two turns of DNA. The nucleosome filament tein–DNA complex fundamental to genome packaging, is then folded into a 30-nm fiber mediated in part by function, and regulation. A fraction of histones are non- nucleosome–nucleosome interactions, and this fiber is allelic variants that have specific expression, localiza- probably the template for most nuclear processes. Addi- tion, and species-distribution patterns. Here we discuss tional levels of compaction enable these fibers to be recent progress in understanding how histone variants packaged into the small volume of the nucleus. lead to changes in chromatin structure and dynamics to The packaging of DNA into nucleosomes and chroma- carry out specific functions. In addition, we review his- tin positively or negatively affects all nuclear processes tone variant assembly into chromatin, the structure of in the cell. While nucleosomes have long been viewed as the variant chromatin, and post-translational modifica- stable entities, there is a large body of evidence indicat- tions that occur on the variants. ing that they are highly dynamic (for review, see Ka- makaka 2003), capable of being altered in their compo- Supplemental material is available at http://www.genesdev.org. sition, structure, and location along the DNA. Enzyme Approximately two meters of human diploid DNA are complexes that either post-translationally modify the packaged into the cell’s nucleus with a volume of ∼1000 histones or alter the position and structure of the nucleo- µm3. -

How Human H1 Histone Recognizes DNA
molecules Article How Human H1 Histone Recognizes DNA Olesya P. Luzhetskaya, Sergey E. Sedykh and Georgy A. Nevinsky * Institute of Chemical Biology and Fundamental Medicine, SD of Russian Academy of Sciences, 8 Lavrentiev Ave., 630090 Novosibirsk, Russia; [email protected] (O.P.L.); [email protected] (S.E.S.) * Correspondence: [email protected]; Tel.: +7-383-363-51-26; Fax: +7-383-363-51-53 Received: 11 August 2020; Accepted: 1 October 2020; Published: 5 October 2020 Abstract: Linker H1 histone is one of the five main histone proteins (H1, H2A, H2B, H3, and H4), which are components of chromatin in eukaryotic cells. Here we have analyzed the patterns of DNA recognition by free H1 histone using a stepwise increase of the ligand complexity method; the affinity of H1 histone for various single- and double-stranded oligonucleotides (d(pN)n; n = 1–20) was evaluated using their competition with 12-mer [32P]labeled oligonucleotide and protein–oligonucleotide complex delaying on nitrocellulose membrane filters. It was shown that minimal ligands of H1 histone (like other DNA-dependent proteins and enzymes) are different mononucleotides (dNMPs; Kd = (1.30 0.2) 2 ± 10 M). An increase in the length of single-stranded (ss) homo- and hetero-oligonucleotides (d(pA)n, × − d(pT)n, d(pC)n, and d(pN)n with different bases) by one nucleotide link regardless of their bases, leads to a monotonic increase in their affinity by a factor of f = 3.0 0.2. This factor f corresponds ± to the Kd value = 1/f characterizing the affinity of one nucleotide of different ss d(pN)n for H1 at n = 2–6 (which are covered by this protein globule) is approximately 0.33 0.02 M. -

The Mechanism of Cross-Talk Between Histone H2B Ubiquitination and H3 Methylation by Dot1l
bioRxiv preprint doi: https://doi.org/10.1101/501098; this version posted December 22, 2018. The copyright holder for this preprint (which was not certified by peer review) is the author/funder, who has granted bioRxiv a license to display the preprint in perpetuity. It is made available under aCC-BY-NC-ND 4.0 International license. The mechanism of cross-talk between histone H2B ubiquitination and H3 methylation by Dot1L Evan J. Worden, Niklas Hoffmann, Chad Hicks and Cynthia Wolberger* Department of Biophysics and Biophysical Chemistry, Johns Hopkins University School of Medicine, Baltimore, MD USA *Corresponding author: [email protected] bioRxiv preprint doi: https://doi.org/10.1101/501098; this version posted December 22, 2018. The copyright holder for this preprint (which was not certified by peer review) is the author/funder, who has granted bioRxiv a license to display the preprint in perpetuity. It is made available under aCC-BY-NC-ND 4.0 International license. Abstract Methylation of histone H3, lysine 79 (H3K79), by Dot1L is a hallmark of actively transcribed genes that depends on monoubiquitination of H2B at lysine 120 (H2B-Ub), and is a well-characterized example of histone modification cross-talk that is conserved from yeast to humans. The mechanism by which H2B-Ub stimulates Dot1L to methylate the relatively inaccessible histone core H3K79 residue is unknown. The 3.0 Å resolution cryo-EM structure of Dot1L bound to ubiquitinated nucleosome reveals that Dot1L contains binding sites for both ubiquitin and the histone H4 tail, which establish two regions of contact that stabilize a catalytically competent state and positions the Dot1L active site over H3K79. -

Combinatorial Genomic Data Refute the Human Chromosome 2 Evolutionary Fusion and Build a Model of Functional Design for Interstitial Telomeric Repeats
The Proceedings of the International Conference on Creationism Volume 8 Print Reference: Pages 222-228 Article 32 2018 Combinatorial Genomic Data Refute the Human Chromosome 2 Evolutionary Fusion and Build a Model of Functional Design for Interstitial Telomeric Repeats Jeffrey P. Tomkins Institute for Creation Research Follow this and additional works at: https://digitalcommons.cedarville.edu/icc_proceedings Part of the Biology Commons, and the Genomics Commons DigitalCommons@Cedarville provides a publication platform for fully open access journals, which means that all articles are available on the Internet to all users immediately upon publication. However, the opinions and sentiments expressed by the authors of articles published in our journals do not necessarily indicate the endorsement or reflect the views of DigitalCommons@Cedarville, the Centennial Library, or Cedarville University and its employees. The authors are solely responsible for the content of their work. Please address questions to [email protected]. Browse the contents of this volume of The Proceedings of the International Conference on Creationism. Recommended Citation Tomkins, J.P. 2018. Combinatorial genomic data refute the human chromosome 2 evolutionary fusion and build a model of functional design for interstitial telomeric repeats. In Proceedings of the Eighth International Conference on Creationism, ed. J.H. Whitmore, pp. 222–228. Pittsburgh, Pennsylvania: Creation Science Fellowship. Tomkins, J.P. 2018. Combinatorial genomic data refute the human chromosome 2 evolutionary fusion and build a model of functional design for interstitial telomeric repeats. In Proceedings of the Eighth International Conference on Creationism, ed. J.H. Whitmore, pp. 222–228. Pittsburgh, Pennsylvania: Creation Science Fellowship. COMBINATORIAL GENOMIC DATA REFUTE THE HUMAN CHROMOSOME 2 EVOLUTIONARY FUSION AND BUILD A MODEL OF FUNCTIONAL DESIGN FOR INTERSTITIAL TELOMERIC REPEATS Jeffrey P. -

Histone H1 Is Essential for Mitotic Chromosome Architecture
Published Online: 20 June, 2005 | Supp Info: http://doi.org/10.1083/jcb.200503031 JCB: ARTICLE Downloaded from jcb.rupress.org on May 12, 2019 Histone H1 is essential for mitotic chromosome architecture and segregation in Xenopus laevis egg extracts Thomas J. Maresca, Benjamin S. Freedman, and Rebecca Heald Department of Molecular and Cell Biology, University of California, Berkeley, Berkeley, CA 94720 uring cell division, condensation and resolution of spindle. Although functional kinetochores assembled, chromosome arms and the assembly of a func- aligned, and exhibited poleward movement, long and D tional kinetochore at the centromere of each sister tangled chromosome arms could not be segregated in chromatid are essential steps for accurate segregation of anaphase. Histone H1 depletion did not significantly affect the genome by the mitotic spindle, yet the contribution of the recruitment of known structural or functional chromo- individual chromatin proteins to these processes is poorly somal components such as condensins or chromokinesins, understood. We have investigated the role of embryonic suggesting that the loss of H1 affects chromosome archi- linker histone H1 during mitosis in Xenopus laevis egg tecture directly. Thus, our results indicate that linker histone extracts. Immunodepletion of histone H1 caused the assem- H1 plays an important role in the structure and function of bly of aberrant elongated chromosomes that extended off vertebrate chromosomes in mitosis. the metaphase plate and outside the perimeter of the Introduction Correct transmission of the genome by the mitotic spindle complexes condensin and cohesin, which are thought to form during cell division requires dramatic changes in chromosome ring structures that generate chromosome super coiling or architecture. -

Epigenetic Control of Mammalian Centromere Protein Binding: Does DNA Methylation Have a Role?
Journal of Cell Science 109, 2199-2206 (1996) 2199 Printed in Great Britain © The Company of Biologists Limited 1996 JCS3386 Epigenetic control of mammalian centromere protein binding: does DNA methylation have a role? Arthur R. Mitchell*, Peter Jeppesen, Linda Nicol†, Harris Morrison and David Kipling MRC Human Genetics Unit, Western General Hospital, Crewe Road, Edinburgh EH4 2XU, UK *Author for correspondence (internet [email protected]) †Present address: MRC Reproductive Biology Unit, Edinburgh, UK SUMMARY Chromosome 1 of the inbred mouse strain DBA/2 has a block of minor satellite DNA sequences on chromosome 1. polymorphism associated with the minor satellite DNA at The binding of the CENP-E protein does not appear to be its centromere. The more terminal block of satellite DNA affected by demethylation of the minor satellite sequences. sequences on this chromosome acts as the centromere as We present a model to explain these observations. This shown by the binding of CREST ACA serum, anti-CENP- model may also indicate the mechanism by which the B and anti-CENP-E polyclonal sera. Demethylation of the CENP-B protein recognises specific sites within the arrays minor satellite DNA sequences accomplished by growing of minor satellite DNA on mouse chromosomes. cells in the presence of the drug 5-aza-2′-deoxycytidine results in a redistribution of the CENP-B protein. This protein now binds to an enlarged area on the more terminal Key words: Centromere satellite DNA, Demethylation, Centromere block and in addition it now binds to the more internal antibody INTRODUCTION A common feature of many mammalian pericentromeric domains is that they contain families of repetitive DNA The centromere of mammalian chromosomes is recognised at sequences (Singer, 1982). -

Gene Knockdown of CENPA Reduces Sphere Forming Ability and Stemness of Glioblastoma Initiating Cells
Neuroepigenetics 7 (2016) 6–18 Contents lists available at ScienceDirect Neuroepigenetics journal homepage: www.elsevier.com/locate/nepig Gene knockdown of CENPA reduces sphere forming ability and stemness of glioblastoma initiating cells Jinan Behnan a,1, Zanina Grieg b,c,1, Mrinal Joel b,c, Ingunn Ramsness c, Biljana Stangeland a,b,⁎ a Department of Molecular Medicine, Institute of Basic Medical Sciences, The Medical Faculty, University of Oslo, Oslo, Norway b Norwegian Center for Stem Cell Research, Department of Immunology and Transfusion Medicine, Oslo University Hospital, Oslo, Norway c Vilhelm Magnus Laboratory for Neurosurgical Research, Institute for Surgical Research and Department of Neurosurgery, Oslo University Hospital, Oslo, Norway article info abstract Article history: CENPA is a centromere-associated variant of histone H3 implicated in numerous malignancies. However, the Received 20 May 2016 role of this protein in glioblastoma (GBM) has not been demonstrated. GBM is one of the most aggressive Received in revised form 23 July 2016 human cancers. GBM initiating cells (GICs), contained within these tumors are deemed to convey Accepted 2 August 2016 characteristics such as invasiveness and resistance to therapy. Therefore, there is a strong rationale for targeting these cells. We investigated the expression of CENPA and other centromeric proteins (CENPs) in Keywords: fi CENPA GICs, GBM and variety of other cell types and tissues. Bioinformatics analysis identi ed the gene signature: fi Centromeric proteins high_CENP(AEFNM)/low_CENP(BCTQ) whose expression correlated with signi cantly worse GBM patient Glioblastoma survival. GBM Knockdown of CENPA reduced sphere forming ability, proliferation and cell viability of GICs. We also Brain tumor detected significant reduction in the expression of stemness marker SOX2 and the proliferation marker Glioblastoma initiating cells and therapeutic Ki67. -

PDF Output of CLIC (Clustering by Inferred Co-Expression)
PDF Output of CLIC (clustering by inferred co-expression) Dataset: Num of genes in input gene set: 13 Total number of genes: 16493 CLIC PDF output has three sections: 1) Overview of Co-Expression Modules (CEMs) Heatmap shows pairwise correlations between all genes in the input query gene set. Red lines shows the partition of input genes into CEMs, ordered by CEM strength. Each row shows one gene, and the brightness of squares indicates its correlations with other genes. Gene symbols are shown at left side and on the top of the heatmap. 2) Details of each CEM and its expansion CEM+ Top panel shows the posterior selection probability (dataset weights) for top GEO series datasets. Bottom panel shows the CEM genes (blue rows) as well as expanded CEM+ genes (green rows). Each column is one GEO series dataset, sorted by their posterior probability of being selected. The brightness of squares indicates the gene's correlations with CEM genes in the corresponding dataset. CEM+ includes genes that co-express with CEM genes in high-weight datasets, measured by LLR score. 3) Details of each GEO series dataset and its expression profile: Top panel shows the detailed information (e.g. title, summary) for the GEO series dataset. Bottom panel shows the background distribution and the expression profile for CEM genes in this dataset. Overview of Co-Expression Modules (CEMs) with Dataset Weighting Scale of average Pearson correlations Num of Genes in Query Geneset: 13. Num of CEMs: 1. 0.0 0.2 0.4 0.6 0.8 1.0 Cenpk Cenph Cenpp Cenpu Cenpn Cenpq Cenpl Apitd1 -
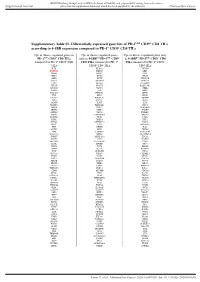
Supplementary Table S5. Differentially Expressed Gene Lists of PD-1High CD39+ CD8 Tils According to 4-1BB Expression Compared to PD-1+ CD39- CD8 Tils
BMJ Publishing Group Limited (BMJ) disclaims all liability and responsibility arising from any reliance Supplemental material placed on this supplemental material which has been supplied by the author(s) J Immunother Cancer Supplementary Table S5. Differentially expressed gene lists of PD-1high CD39+ CD8 TILs according to 4-1BB expression compared to PD-1+ CD39- CD8 TILs Up- or down- regulated genes in Up- or down- regulated genes Up- or down- regulated genes only PD-1high CD39+ CD8 TILs only in 4-1BBneg PD-1high CD39+ in 4-1BBpos PD-1high CD39+ CD8 compared to PD-1+ CD39- CD8 CD8 TILs compared to PD-1+ TILs compared to PD-1+ CD39- TILs CD39- CD8 TILs CD8 TILs IL7R KLRG1 TNFSF4 ENTPD1 DHRS3 LEF1 ITGA5 MKI67 PZP KLF3 RYR2 SIK1B ANK3 LYST PPP1R3B ETV1 ADAM28 H2AC13 CCR7 GFOD1 RASGRP2 ITGAX MAST4 RAD51AP1 MYO1E CLCF1 NEBL S1PR5 VCL MPP7 MS4A6A PHLDB1 GFPT2 TNF RPL3 SPRY4 VCAM1 B4GALT5 TIPARP TNS3 PDCD1 POLQ AKAP5 IL6ST LY9 PLXND1 PLEKHA1 NEU1 DGKH SPRY2 PLEKHG3 IKZF4 MTX3 PARK7 ATP8B4 SYT11 PTGER4 SORL1 RAB11FIP5 BRCA1 MAP4K3 NCR1 CCR4 S1PR1 PDE8A IFIT2 EPHA4 ARHGEF12 PAICS PELI2 LAT2 GPRASP1 TTN RPLP0 IL4I1 AUTS2 RPS3 CDCA3 NHS LONRF2 CDC42EP3 SLCO3A1 RRM2 ADAMTSL4 INPP5F ARHGAP31 ESCO2 ADRB2 CSF1 WDHD1 GOLIM4 CDK5RAP1 CD69 GLUL HJURP SHC4 GNLY TTC9 HELLS DPP4 IL23A PITPNC1 TOX ARHGEF9 EXO1 SLC4A4 CKAP4 CARMIL3 NHSL2 DZIP3 GINS1 FUT8 UBASH3B CDCA5 PDE7B SOGA1 CDC45 NR3C2 TRIB1 KIF14 TRAF5 LIMS1 PPP1R2C TNFRSF9 KLRC2 POLA1 CD80 ATP10D CDCA8 SETD7 IER2 PATL2 CCDC141 CD84 HSPA6 CYB561 MPHOSPH9 CLSPN KLRC1 PTMS SCML4 ZBTB10 CCL3 CA5B PIP5K1B WNT9A CCNH GEM IL18RAP GGH SARDH B3GNT7 C13orf46 SBF2 IKZF3 ZMAT1 TCF7 NECTIN1 H3C7 FOS PAG1 HECA SLC4A10 SLC35G2 PER1 P2RY1 NFKBIA WDR76 PLAUR KDM1A H1-5 TSHZ2 FAM102B HMMR GPR132 CCRL2 PARP8 A2M ST8SIA1 NUF2 IL5RA RBPMS UBE2T USP53 EEF1A1 PLAC8 LGR6 TMEM123 NEK2 SNAP47 PTGIS SH2B3 P2RY8 S100PBP PLEKHA7 CLNK CRIM1 MGAT5 YBX3 TP53INP1 DTL CFH FEZ1 MYB FRMD4B TSPAN5 STIL ITGA2 GOLGA6L10 MYBL2 AHI1 CAND2 GZMB RBPJ PELI1 HSPA1B KCNK5 GOLGA6L9 TICRR TPRG1 UBE2C AURKA Leem G, et al.