Standards and Tools for Model Exchange and Analysis in Systems Biology
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-
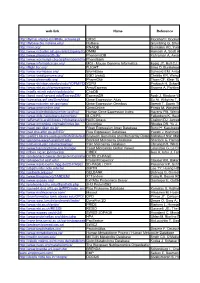
Web-Link Name Reference DRSC Flockhart I, Booker M, Kiger A, Et Al.: Flyrnai: the Drosophila Rnai Screening Center Database
web-link Name Reference http://flyrnai.org/cgi-bin/RNAi_screens.pl DRSC Flockhart I, Booker M, Kiger A, et al.: FlyRNAi: the Drosophila RNAi screening center database. Nucleic Acids Res. 34(Database issue):D489-94 (2006) http://flybase.bio.indiana.edu/ FlyBase Grumbling G, Strelets V.: FlyBase: anatomical data, images and queries. Nucleic Acids Res. 34(Database issue):D484-8 (2006) http://rnai.org/ RNAiDB Gunsalus KC, Yueh WC, MacMenamin P, et al.: RNAiDB and PhenoBlast: web tools for genome-wide phenotypic mapping projects. Nucleic Acids Res. 32(Database issue):D406-10 (2004) http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?db=OMIMOMIM Hamosh A, Scott AF, Amberger JS, et al.: Online Mendelian Inheritance in Man (OMIM), a knowledgebase of human genes and genetic disorders. Nucleic Acids Res. 33(Database issue):D514-7 (2005) http://www.phenomicdb.de/ PhenomicDB Kahraman A, Avramov A, Nashev LG, et al.: PhenomicDB: a multi-species genotype/phenotype database for comparative phenomics. Bioinformatics 21(3):418-20 (2005) http://www.worm.mpi-cbg.de/phenobank2/cgi-bin/MenuPage.pyPhenoBank http://www.informatics.jax.org/ MGI - Mouse Genome Informatics Eppig JT, Bult CJ, Kadin JA, et al.: The Mouse Genome Database (MGD): from genes to mice--a community resource for mouse biology. Nucleic Acids Res. 33(Database issue):D471-5 (2005) http://flight.licr.org/ FLIGHT Sims D, Bursteinas B, Gao Q, et al.: FLIGHT: database and tools for the integration and cross-correlation of large-scale RNAi phenotypic datasets. Nucleic Acids Res. 34(Database issue):D479-83 (2006) http://www.wormbase.org/ WormBase Schwarz EM, Antoshechkin I, Bastiani C, et al.: WormBase: better software, richer content. -

Mendel® SBML Editor User Guide Table of Contents
Mendel® SBML Editor User Guide Table of Contents Introduction. 1 Mendel® SBML Editor overview . 1 Mendel® SBML Editor features . 1 Getting started . 2 Installation notes . 2 Basic tutorial: Describing Reaction Network of Dimerization . 3 Tasks . 13 Navigating the workbench . 13 Basics. 14 Open existing SBML Model . 14 New SBML Model. 15 Save SBML Model . 15 Save SBML Model graph as image. 15 Adding and configuring Nodes . 15 Adding a Species, Reaction or Compartment . 16 Configuring a Species, Reaction or Compartment. 16 Adding and configuring Relations . 17 Adding a Reactant, Modifier or Product . 17 Configuring a Reactant, Modifier or Product . 18 Adding and configuring Kinetic Laws . 18 Adding and configuring Function Definitions . 19 Adding and configuring Unit Definitions. 19 Adding and configuring Parameters . 20 Adding and configuring Rules, Initial Assignments and Constraints . 20 Adding and configuring Events . 21 Adding Notes . 22 Deleting elements. 22 Reference . 24 Graphical User Interface overview . 24 Menu Bar . 24 File Menu Actions . 24 Edit Menu Actions . 25 Diagram Menu Actions. 26 Window Menu Actions. 27 Help Menu Actions . 27 Toolbar . 28 Model outline tab . 29 Graphical overview tab . 30 Graphical Editor tab . 31 Properties tab . 32 Nodes . 32 Species . 32 Reactions . 33 Compartment . 33 Relations . 34 Kinetic Laws . 34 Function Definitions . 34 Unit Definitions . 35 Parameters . 35 Initial Assignments . 35 Rules . 36 Algebraic Rules . .. -

A Bayesian Inference Transcription Factor Activity Model for the Analysis of Single-Cell Transcriptomes
Downloaded from genome.cshlp.org on October 7, 2021 - Published by Cold Spring Harbor Laboratory Press Method A Bayesian inference transcription factor activity model for the analysis of single-cell transcriptomes Shang Gao,1,2,3 Yang Dai,1 and Jalees Rehman1,2,3,4 1Department of Bioengineering, 2Department of Medicine, Division of Cardiology, 3Department of Pharmacology and Regenerative Medicine, University of Illinois at Chicago, Chicago, Illinois 60612, USA; 4University of Illinois Cancer Center, Chicago, Illinois 60612, USA Single-cell RNA sequencing (scRNA-seq) has emerged as a powerful experimental approach to study cellular heterogeneity. One of the challenges in scRNA-seq data analysis is integrating different types of biological data to consistently recognize discrete biological functions and regulatory mechanisms of cells, such as transcription factor activities and gene regulatory networks in distinct cell populations. We have developed an approach to infer transcription factor activities from scRNA-seq data that leverages existing biological data on transcription factor binding sites. The Bayesian inference tran- scription factor activity model (BITFAM) integrates ChIP-seq transcription factor binding information into scRNA-seq data analysis. We show that the inferred transcription factor activities for key cell types identify regulatory transcription factors that are known to mechanistically control cell function and cell fate. The BITFAM approach not only identifies bio- logically meaningful transcription factor activities, -

Data Management in Systems Biology I
Data management in systems biology I – Overview and bibliography Gerhard Mayer, University of Stuttgart, Institute of Biochemical Engineering (IBVT), Allmandring 31, D-70569 Stuttgart Abstract Large systems biology projects can encompass several workgroups often located in different countries. An overview about existing data standards in systems biology and the management, storage, exchange and integration of the generated data in large distributed research projects is given, the pros and cons of the different approaches are illustrated from a practical point of view, the existing software – open source as well as commercial - and the relevant literature is extensively overviewed, so that the reader should be enabled to decide which data management approach is the best suited for his special needs. An emphasis is laid on the use of workflow systems and of TAB-based formats. The data in this format can be viewed and edited easily using spreadsheet programs which are familiar to the working experimental biologists. The use of workflows for the standardized access to data in either own or publicly available databanks and the standardization of operation procedures is presented. The use of ontologies and semantic web technologies for data management will be discussed in a further paper. Keywords: MIBBI; data standards; data management; data integration; databases; TAB-based formats; workflows; Open Data INTRODUCTION the foundation of a new journal about biological The large amount of data produced by biological databases [24], the foundation of the ISB research projects grows at a fast rate. The 2009 (International Society for Biocuration) and special edition of the annual Nucleic Acids Research conferences like DILS (Data Integration in the Life database issue mentions 1170 databases [1]; alone Sciences) [25]. -

Tellurium: a Python Based Modeling and Reproducibility Platform for Systems Biology
bioRxiv preprint doi: https://doi.org/10.1101/054601; this version posted June 2, 2016. The copyright holder for this preprint (which was not certified by peer review) is the author/funder, who has granted bioRxiv a license to display the preprint in perpetuity. It is made available under aCC-BY 4.0 International license. Tellurium: A Python Based Modeling and Reproducibility Platform for Systems Biology Kiri Choi ([email protected]),∗†‡ J. Kyle Medley ([email protected]),†‡ Caroline Cannistra ([email protected]),‡ Matthias K¨onig([email protected]),§ Lucian Smith ([email protected]),‡ Kaylene Stocking ([email protected]),‡ and Herbert M. Sauro ([email protected])‡ ‡Department of Bioengineering, University of Washington, Seattle, WA 98195, USA Address: Department of Bioengineering, University of Washington, Box 355061, Seattle, WA 98195-5061, USA; Tel: (+1) 206 685 2119 §Institute for Theoretical Biology, Humboldt University of Berlin, Berlin, Germany Address : Humboldt-University Berlin, Institute for Theoretical Biology Invalidenstraße 43, 10115 Berlin, Germany; Tel: (+49) 30 20938450 keyword: systems biology, reproducibility, exchangeability, simulations, software tools May 20, 2016 ∗To whom correspondence should be addressed †These authors contributed equally to this work 1 bioRxiv preprint doi: https://doi.org/10.1101/054601; this version posted June 2, 2016. The copyright holder for this preprint (which was not certified by peer review) is the author/funder, who has granted bioRxiv a license to display the preprint in perpetuity. It is made available under aCC-BY 4.0 International license. Abstract In this article, we present Tellurium, a powerful Python-based integrated environment de- signed for model building, analysis, simulation and reproducibility in systems and synthetic biology. -

COPASI Documentation VERSION 4.3 (BUILD 25) 1 / 113
COPASI Documentation VERSION 4.3 (BUILD 25) 1 / 113 COPASI Documentation Version 4.3 (Build 25) COPASI Development Team on April 3, 2008 COPASI Documentation VERSION 4.3 (BUILD 25) 2 / 113 COPASI Documentation VERSION 4.3 (BUILD 25) 3 / 113 Contents 1 Model Creation 8 1.1 Introduction . 8 1.2 Commandline Version and Commandline Options . 8 1.3 COPASI GUI Elements . 9 1.4 General Model Settings . 10 1.5 Compartments . 12 1.6 Species . 16 1.7 Reactions . 20 1.8 Global Quantities . 22 1.9 Parameter View . 24 1.10 User Defined Functions . 25 1.11 Sliders . 29 1.12 Tutorial Wizard . 32 2 Output 33 2.1 Predefined Reports . 33 2.2 Output Assistant . 33 2.3 Manual Definition . 34 2.3.1 Reports . 34 2.3.2 Plots . 37 3 Simple Tasks 40 3.1 Steady-State Analysis . 40 3.2 Stoichiometric State Analysis . 41 3.2.1 Elementary Flux Modes . 41 3.2.2 Mass Conservations . 42 3.3 Time Course Simulation . 43 3.3.1 Working with Plots . 45 3.4 Metabolic Control Analysis (MCA) . 46 3.5 Lyapunov Exponents . 47 COPASI Documentation VERSION 4.3 (BUILD 25) 4 / 113 4 Complex Tasks 49 4.1 Parameter Scan . 49 4.2 Optimization . 55 4.3 Parameter Estimation . 57 4.3.1 Experimental Data . 58 4.3.2 Result . 60 4.4 Sensitivity Analysis . 61 5 Importing and Exporting 62 5.1 Importing and Exporting SBML files . 62 5.2 Exporting C Source files . 63 5.3 Exporting Berkeley Madonna files . 65 5.4 Exporting XPPAUT files . -
A Modular Mathematical Model of Exercise-Induced Changes In
bioRxiv preprint doi: https://doi.org/10.1101/2021.05.31.446385; this version posted May 31, 2021. The copyright holder for this preprint (which was not certified by peer review) is the author/funder. All rights reserved. No reuse allowed without permission. 1 A modular mathematical model of 2 exercise-induced changes in metabolism, 3 signaling, and gene expression in human 4 skeletal muscle 5 6 Akberdin I.R.1,2,3,4, Kiselev I.N.1,4,5, Pintus S.S.1,4,5, Sharipov, R.N.1,3,4,5, Vertyshev A.Yu.6, 7 Vinogradova O.L.7, Popov D.V.7, Kolpakov F.A.1,4,5 8 9 1 BIOSOFT.RU, LLC, Novosibirsk, Russian Federation, [email protected] 10 2 Federal Research Center Institute of Cytology and Genetics SB RAS, Novosibirsk, Russia 11 3 Novosibirsk State University, Novosibirsk, Russia 12 4 Sirius University of Science and Technology, Sochi, Russia 13 5 Federal Research Center for Information and Computational Technologies, Novosibirsk, Russia5 14 6 CJSC "Sites-Tsentr", Moscow, Russia 15 7 Institute of Biomedical Problems of the Russian Academy of Sciences, Moscow, Russia 16 17 Abstract 18 Skeletal muscle is the principal contributor to exercise-induced changes in human 19 metabolism. Strikingly, although it has been demonstrated that a lot of 20 metabolites accumulating in blood and human skeletal muscle during an exercise 21 activate different signaling pathways and induce expression of many genes in 22 working muscle fibres, the system understanding of signaling-metabolic 23 pathways interrelations with downstream genetic regulation in the skeletal 24 muscle is still elusive. -
CYCLONET—An Integrated Database on Cell Cycle Regulation And
D550–D556 Nucleic Acids Research, 2007, Vol. 35, Database issue doi:10.1093/nar/gkl912 CYCLONET—an integrated database on cell cycle regulation and carcinogenesis Fedor Kolpakov1,2, Vladimir Poroikov3, Ruslan Sharipov1,2,4,*, Yury Kondrakhin1,2, Alexey Zakharov3, Alexey Lagunin3, Luciano Milanesi5 and Alexander Kel6 1Institute of Systems Biology, 15, Detskiy proezd, Novosibirsk 630090, Russia, 2Design Technological Institute of Digital Techniques, Siberian Branch of Russian Academy of Sciences, 6, Institutskaya, Novosibirsk 630090, Russia, 3Institute of Biomedical Chemistry of Russian Academy of Medical Sciences, 10, 4 Pogodinskaya Street, Moscow 119121, Russia, Institute of Cytology and Genetics, Siberian Branch of Downloaded from Russian Academy of Sciences, 10, Lavrentyev Avenue, Novosibirsk 630090, Russia, 5CNR-Institute of Biomedical Technologies, 93, Via Fratelli Cervi, Segrate (MI) 20090, Italy and 6BIOBASE GmbH, 33, Halchtersche Strasse, Wolfenbuettel 38304, Germany Received August 16, 2006; Revised October 12, 2006; Accepted October 13, 2006 http://nar.oxfordjournals.org/ ABSTRACT INTRODUCTION Computational modelling of mammalian cell cycle The main goal of the Cyclonet database is to integrate information from genomics, proteomics, chemoinformatics regulation is a challenging task, which requires at McGill University Libraries on December 28, 2012 comprehensive knowledge on many interrelated and systems biology on mammalian cell cycle regulation in processes in the cell. We have developed a web- normal and pathological states. -
View
Boyarskikh et al. BMC Medical Genomics 2018, 11(Suppl 1):12 DOI 10.1186/s12920-018-0330-5 RESEARCH Open Access Computational master-regulator search reveals mTOR and PI3K pathways responsible for low sensitivity of NCI-H292 and A427 lung cancer cell lines to cytotoxic action of p53 activator Nutlin-3 Ulyana Boyarskikh1, Sergey Pintus2, Nikita Mandrik2, Daria Stelmashenko2, Ilya Kiselev2, Ivan Evshin2, Ruslan Sharipov2, Philip Stegmaier3, Fedor Kolpakov2, Maxim Filipenko1 and Alexander Kel1,2,3* From Belyaev Conference Novosibirsk, Russia. 07-10 August 2017 Abstract Background: Small molecule Nutlin-3 reactivates p53 in cancer cells by interacting with the complex between p53 and its repressor Mdm-2 and causing an increase in cancer cell apoptosis. Therefore, Nutlin-3 has potent anticancer properties. Clinical and experimental studies of Nutlin-3 showed that some cancer cells may lose sensitivity to this compound. Here we analyze possible mechanisms for insensitivity of cancer cells to Nutlin-3. Methods: We applied upstream analysis approach implemented in geneXplain platform (genexplain.com) using TRANSFAC® database of transcription factors and their binding sites in genome and using TRANSPATH® database of signal transduction network with associated software such as Match™ and Composite Module Analyst (CMA). Results: Using genome-wide gene expression profiling we compared several lung cancer cell lines and showed that expression programs executed in Nutlin-3 insensitive cell lines significantly differ from that of Nutlin-3 sensitive cell lines. Using artificial intelligence approach embed in CMA software, we identified a set of transcription factors cooperatively binding to the promoters of genes up-regulated in the Nutlin-3 insensitive cell lines. -

Comprehensive Modeling Platform for Photosynthetic Organisms
MASARYK UNIVERSITY FACULTY OF INFORMATICS COMPREHENSIVE MODELING PLATFORM FOR PHOTOSYNTHETIC ORGANISMS THESIS MATEJ KLEMENT 2012 Contents 1 Introduction4 1.1 Objectives..................................5 2 State of the art6 2.1 Data Exchange formats..........................8 2.1.1 SBML................................8 2.1.2 CellML................................9 2.1.3 BioPAX................................9 2.1.4 PSI-MI................................ 10 2.1.5 SBGN................................ 10 2.1.6 Format of Matlab.......................... 11 2.1.7 Format Octave........................... 11 2.2 Data Exchange and modeling tools................... 11 2.2.1 Biomodels.net........................... 12 2.2.2 CellML.org.............................. 13 2.2.3 Copasi................................ 13 2.2.4 Vcell................................. 13 2.2.5 E-cell................................. 14 2.2.6 ProMot................................ 14 2.2.7 PaxTools............................... 15 2.2.8 Matlab................................ 15 2.2.9 Octave................................ 16 2.2.10Scilab................................ 16 2.2.11BioUML............................... 16 2.3 Annotation ontology............................ 17 2.3.1 Gene Ontology........................... 17 2.3.2 KEGG................................ 17 2.3.3 SBO................................. 17 2.4 Photosynthesis modeling......................... 17 3 Aims 18 3.1 Theoretical aims.............................. 18 3.2 Practical aims.............................. -

Synthetic Animal: Trends in Animal Breeding and Genetics
Open Access Insights in Biology and Medicine Review Article Synthetic Animal: Trends in Animal Breeding and Genetics ISSN 1 2 2639-6769 Abolfazl Bahrami * and Ali Najafi 1Department of Animal Science, University College of Agriculture and Natural Resources, University of Tehran, Karaj, Iran 2Molecular Biology Research Center, Baqiyatallah University of Medical Sciences, Tehran, Iran *Address for Correspondence: A Bahrami, Abstract Department of Animal Science, Tehran University, Karaj, I.R. Iran, Tel/Fax: +98 9199300065; Email: Synthetic biology is an interdisciplinary branch of biology and engineering. The subject [email protected] combines various disciplines from within these domains, such as biotechnology, evolutionary Submitted: 31 December 2018 biology, molecular biology, systems biology, biophysics, computer engineering, and genetic Approved: 10 January 2019 engineering. Synthetic biology aims to understand whole biological systems working as a unit, Published: 11 January 2019 rather than investigating their individual components and design new genome. Signifi cant advances have been made using systems biology and synthetic biology approaches, especially in the fi eld Copyright: © 2018 Bahrami A, et al. This is of bacterial and eukaryotic cells. Similarly, progress is being made with ‘synthetic approaches’ in an open access article distributed under the genetics and animal sciences, providing exciting opportunities to modulate, genome design and Creative Commons Attribution License, which permits unrestricted use, distribution, and fi nally synthesis animal for favorite traits. reproduction in any medium, provided the original work is properly cited Keywords: Synthetic biology; Systems biology; Introduction Synthetic approaches; Genetic engineering Animal breeding In 1859, Charles Darwin published his book ‘On the origin of species’, based on the indings that he collected during his voyage on ‘the Beagle’ [1]. -

COPASI Documentation I
COPASI Documentation i COPASI Documentation Version 4.6 (Build 32) COPASI Development Team on August 3, 2010 COPASI Documentation 2 / 131 COPASI Documentation 3 / 131 Contents 1 Model Creation 1 1.1 Introduction . 1 1.2 Commandline Version and Commandline Options . 1 1.3 COPASI GUI Elements . 3 1.4 General Model Settings . 3 1.5 Compartments . 5 1.6 Species . 10 1.7 Reactions . 13 1.8 Global Quantities . 15 1.9 Events . 17 1.9.1 Trigger . 17 1.9.2 Assignment . 17 1.9.3 Delay . 18 1.10 Annotating Models and Model Elements . 19 1.11 Parameter View . 20 1.12 User Defined Functions . 21 1.12.1 Standard Operators . 23 1.12.2 Miscellaneaous Functions . 23 1.12.3 Trigonometric Functions . 24 1.12.4 Random Distribuitions . 24 1.12.5 Logical Operators . 24 1.12.6 Conditional Statement . 25 1.12.7 Parenthesis . 25 1.12.8 Built-in Constants . 25 1.13 Sliders . 26 1.14 Tutorial Wizard . 28 2 Output 30 2.1 Predefined Reports . 30 2.2 Output Assistant . 30 2.3 Manual Definition . 31 2.3.1 Reports . 31 2.3.2 Plots . 34 COPASI Documentation 4 / 131 3 Tasks 37 3.1 Steady-State Analysis . 37 3.2 Stoichiometric State Analysis . 38 3.2.1 Elementary Flux Modes . 38 3.2.2 Mass Conservations . 39 3.3 Time Course Simulation . 40 3.3.1 Working with Plots . 42 3.4 Metabolic Control Analysis (MCA) . 43 3.5 Lyapunov Exponents . 44 3.6 Time Scale Separation . 45 3.7 Parameter Scan .