Screening of HBB Gene Mutations in Population Samples from Alentejo and Implementation of a ® Snapshot Based System for HBB*S Haplotyping
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

Supporting Information
Supporting Information Biagioli et al. 10.1073/pnas.0813216106 SI Text and the procedure was repeated twice. Single cells were then RNA Isolation, Reverse Transcription, qPCR, Cloning, and Sequencing. resuspended in panning buffer and incubated on lectin-coated RNA was extracted from cell lines and blood by using TRIzol panning plates for 15 min at room temperature. Nonadherent reagent (Invitrogen) and following vendor instructions. cells were transferred to the next panning plate (four pannings, RNA was extracted from LCM- or FACS-purified cells with an 15 min each). Then, nonadherent cells were collected, centri- Absolutely RNA Nanoprep Kit (Stratagene). Single-strand fuged, and resuspended in serum-free neuronal medium or PBS. cDNA was obtained from purified RNA by using the iSCRIPT A similar procedure was also followed for the dissociation of cDNA Synhesis Kit (Bio-Rad) according to the manufacturer’s cortical and hippocampal astrocytes and oligodendrocytes. instructions. Quantitative RT-PCR was performed by using A cell strainer with 70-m nylon mesh was used to obtain a SYBER-Green PCR Master Mix and an iQ5 Real-Time PCR single-cell suspension (BD Falcon) before sorting. 7-AAD Detection System (Bio-Rad). (Beckman–Coulter) was added to the cell suspension to exclude Quantitative RT-PCR was performed with an iCycler IQ dead cells. A high-speed cell sorter (MoFlo) was used to sort (Bio-Rad); -actin was used as an endogenous control to nor- subpopulation of cells expressing GFP. Sorting parameters used malize the expression level of target genes. Primers were chosen for the three different populations are visualized in Fig. -
Technical Note, Appendix: an Analysis of Blood Processing Methods to Prepare Samples for Genechip® Expression Profiling (Pdf, 1
Appendix 1: Signature genes for different blood cell types. Blood Cell Type Source Probe Set Description Symbol Blood Cell Type Source Probe Set Description Symbol Fraction ID Fraction ID Mono- Lympho- GSK 203547_at CD4 antigen (p55) CD4 Whitney et al. 209813_x_at T cell receptor TRG nuclear cytes gamma locus cells Whitney et al. 209995_s_at T-cell leukemia/ TCL1A Whitney et al. 203104_at colony stimulating CSF1R lymphoma 1A factor 1 receptor, Whitney et al. 210164_at granzyme B GZMB formerly McDonough (granzyme 2, feline sarcoma viral cytotoxic T-lymphocyte- (v-fms) oncogene associated serine homolog esterase 1) Whitney et al. 203290_at major histocompatibility HLA-DQA1 Whitney et al. 210321_at similar to granzyme B CTLA1 complex, class II, (granzyme 2, cytotoxic DQ alpha 1 T-lymphocyte-associated Whitney et al. 203413_at NEL-like 2 (chicken) NELL2 serine esterase 1) Whitney et al. 203828_s_at natural killer cell NK4 (H. sapiens) transcript 4 Whitney et al. 212827_at immunoglobulin heavy IGHM Whitney et al. 203932_at major histocompatibility HLA-DMB constant mu complex, class II, Whitney et al. 212998_x_at major histocompatibility HLA-DQB1 DM beta complex, class II, Whitney et al. 204655_at chemokine (C-C motif) CCL5 DQ beta 1 ligand 5 Whitney et al. 212999_x_at major histocompatibility HLA-DQB Whitney et al. 204661_at CDW52 antigen CDW52 complex, class II, (CAMPATH-1 antigen) DQ beta 1 Whitney et al. 205049_s_at CD79A antigen CD79A Whitney et al. 213193_x_at T cell receptor beta locus TRB (immunoglobulin- Whitney et al. 213425_at Homo sapiens cDNA associated alpha) FLJ11441 fis, clone Whitney et al. 205291_at interleukin 2 receptor, IL2RB HEMBA1001323, beta mRNA sequence Whitney et al. -

Recombinant Human Hemoglobin Subunit Theta-1/HBQ1 (N-6His)
9853 Pacific Heights Blvd. Suite D. San Diego, CA 92121, USA Tel: 858-263-4982 Email: [email protected] 32-8592: Recombinant Human Hemoglobin Subunit theta-1/HBQ1 (N-6His) Gene : HBQ1 Gene ID : 3049 Uniprot ID : P09105 Description Source: E.coli. MW :17.7kD. Recombinant Human Hemoglobin subunit theta-1 is produced by our E.coli expression system and the target gene encoding Met1-Arg142 is expressed with a 6His tag at the N-terminus. Hemoglobin subunit theta-1 is a protein that in humans is encoded by the HBQ1 gene. Theta-globin mRNA is originally found in human fetal erythroid tissue but not in adult erythroid or other nonerythroid tissue. Theta-1 is a member of the human alpha-globin gene cluster that includes five functional genes and two pseudogenes. Research supports a transcriptionally active role for the gene and a functional role for the peptide in specific cells, possibly those of early erythroid tissue. Hemoglobin has a quaternary structure characteristically composed of many multi- subunit globular proteins. Most of the amino acids in hemoglobin form alpha helices, connected by short non-helical segments. Hydrogen bonds stabilize the helical sections inside this protein, causing attractions within the molecule, folding each polypeptide chain into a specific shape. Hemoglobin's quaternary structure comes from its four subunits in roughly a tetrahedral arrangement. Product Info Amount : 10 µg / 50 µg Content : Lyophilized from a 0.2 µm filtered solution of 20mM PB, 150mM NaCl, pH 7.0. Lyophilized protein should be stored at -20°C, though stable at room temperature for 3 weeks. -

Analysis of Genetic Determinants Associated with Persistent Synthesis of Fetal Hemoglobin
UNIVERSITÀ DEGLI STUDI DI SASSARI PhD School in Biomolecular and Biotechnological Sciences Curriculum: Biochemistry and Molecular Biology Director: Prof. Claudia Crosio “XXVI Ciclo” Analysis of genetic determinants associated with Persistent Synthesis of Fetal Hemoglobin Supervisor: Monica Pirastru, PhD Director: Prof. Claudia Crosio PhD Student: Sandro Trova ................................................................................................................................................. INDEX INDEX ABSTRACT ................................................................................... 3 INTRODUCTION ......................................................................... 4 1. Hemoglobin .......................................................................................... 4 1.1 Structure and function of Hemoglobin ........................................ 4 1.2 Structure of globin genes and their cluster organization ............. 5 1.3 Genomic context of the α– and β–globin gene clusters .............. 9 2. Globin gene switching ....................................................................... 12 2.1 Regulatory regions and transcription factors of globin genes ... 13 2.2 The β–Globin Locus Control Region (β–LCR) role in globin expression ....................................................................... 20 2.3 Chromatin role in β–like globin gene expression: the PYR role .............................................................................. 25 2.4 Summary on the fetal to adult switch ....................................... -

Simultaneous Detection of Target Cnvs and Snvs of Thalassemia by Multiplex PCR and Next‑Generation Sequencing
MOLECULAR MEDICINE REPORTS 19: 2837-2848, 2019 Simultaneous detection of target CNVs and SNVs of thalassemia by multiplex PCR and next‑generation sequencing DONG-MEI FAN1*, XU YANG2*, LI-MIN HUANG1, GUO-JUN OUYANG3, XUE-XI YANG1 and MING LI1 1Institute of Antibody Engineering, School of Laboratory Medicine and Biotechnology, Southern Medical University, Guangzhou, Guangdong 510515; 2Clinical Innovation and Research Center, Shenzhen Hospital of Southern Medical University, Shenzhen, Guangdong 518110; 3Guangzhou Darui Biotechnology Co., Ltd., Guangzhou, Guangdong 510663, P.R. China Received May 16, 2018; Accepted December 3, 2018 DOI: 10.3892/mmr.2019.9896 Abstract. Thalassemia is caused by complex mechanisms, Introduction including copy number variants (CNVs) and single nucleotide variants (SNVs). The CNV types of α-thalassemia are typi- Thalassemia is caused by copy number variants (CNVs) and cally detected by gap-polymerase chain reaction (PCR). The single nucleotide variants (SNVs) in the α-globin (HBA) or SNV types are detected by Sanger sequencing. In the present β-globin (HBB) genes that result in the absence or lack of α- or study, a novel method was developed that simultaneously β-globin chains, and ultimately hemolytic anemia. It is esti- detects CNVs and SNVs by multiplex PCR and next-genera- mated that ~7% of the world population carries the gene for the tion sequencing (NGS). To detect CNVs, 33 normal samples disease (1), and the birth rate of children with hemoglobin (Hb) were used as a cluster of control values to build a baseline, disorders is ≥2.4% per year (2). Thalassemia occurs most in the and the A, B, C, and D ratios were developed to evaluate-SEA, Mediterranean region, East South Asia, and the subcontinents -α4.2, -α3.7, and compound or homozygous CNVs, respectively. -

Normal, C=Critical
Patient Report |FINAL Client: Example Client ABC123 Patient: Patient, Example 123 Test Drive Salt Lake City, UT 84108 DOB 7/22/1970 UNITED STATES Gender: Female Patient Identifiers: 01234567890ABCD, 012345 Physician: Doctor, Example Visit Number (FIN): 01234567890ABCD Collection Date: 00/00/0000 00:00 Hemoglobin Evaluation Reflexive Cascade ARUP test code 2005792 Hemoglobin A 97.1 % (Ref Interval: 95.0-97.9) Hemoglobin A2 2.6 % (Ref Interval: 2.0-3.5) Hemoglobin F 0.3 % (Ref Interval: 0.0-2.1) REFERENCE INTERVAL: Hemoglobin F Access complete set of age- and/or gender-specific reference intervals for this test in the ARUP Laboratory Test Directory (aruplab.com). Hemoglobin S 0.0 % (Ref Interval: 0.0-0.0) Hemoglobin C 0.0 % (Ref Interval: 0.0-0.0) Hemoglobin E 0.0 % (Ref Interval: 0.0-0.0) Hemoglobin - Other 0.0 % (Ref Interval: 0.0-0.0) Sickle Cell Solubility Not Performed Hemoglobin, Capillary Electrophoresis Performed Hemoglobin Evaluation See Note Beta Globin Full Gene Sequencing Not Applicable H=High, L=Low, *=Abnormal, C=Critical Patient: Patient, Example ARUP Accession: 21-048-400496 Patient Identifiers: 01234567890ABCD, 012345 Visit Number (FIN): 01234567890ABCD Page 1 of 4 | Printed: 3/11/2021 11:41:09 AM 4848 Patient Report |FINAL Beta Globin (HBB) Del/Dup Result Not Applicable Alpha Thalassemia HBA1 and HBA2 Seq Not Applicable Gamma Globin (HBG1 and HBG2) Sequencing Not Applicable Hemoglobin Cascade Interpretation See Note H=High, L=Low, *=Abnormal, C=Critical Patient: Patient, Example ARUP Accession: 21-048-400496 Patient Identifiers: 01234567890ABCD, 012345 Visit Number (FIN): 01234567890ABCD Page 2 of 4 | Printed: 3/11/2021 11:41:09 AM 4848 Patient Report |FINAL RESULT Normal hemoglobin evaluation. -

Globin Variants Modify Hematologic and Other Clinical Phenotypes in Sickle Cell Trait and Disease
Common α-globin variants modify hematologic and other clinical phenotypes in sickle cell trait and disease The Harvard community has made this article openly available. Please share how this access benefits you. Your story matters Citation Raffield, L. M., J. C. Ulirsch, R. P. Naik, S. Lessard, R. E. Handsaker, D. Jain, H. M. Kang, et al. 2018. “Common α-globin variants modify hematologic and other clinical phenotypes in sickle cell trait and disease.” PLoS Genetics 14 (3): e1007293. doi:10.1371/journal.pgen.1007293. http://dx.doi.org/10.1371/ journal.pgen.1007293. Published Version doi:10.1371/journal.pgen.1007293 Citable link http://nrs.harvard.edu/urn-3:HUL.InstRepos:37068183 Terms of Use This article was downloaded from Harvard University’s DASH repository, and is made available under the terms and conditions applicable to Other Posted Material, as set forth at http:// nrs.harvard.edu/urn-3:HUL.InstRepos:dash.current.terms-of- use#LAA RESEARCH ARTICLE Common α-globin variants modify hematologic and other clinical phenotypes in sickle cell trait and disease Laura M. Raffield1☯, Jacob C. Ulirsch2,3,4☯, Rakhi P. Naik5☯, Samuel Lessard6,7, Robert E. Handsaker4,8,9, Deepti Jain10, Hyun M. Kang11, Nathan Pankratz12, Paul L. Auer13, Erik L. Bao2,3,4, Joshua D. Smith14, Leslie A. Lange15, Ethan M. Lange15, Yun Li1,16,17, Timothy A. Thornton11, Bessie A. Young18,19, Goncalo R. Abecasis20, Cathy C. Laurie10, Deborah A. Nickerson14, Steven A. McCarroll4,8,9, Adolfo Correa21, James G. Wilson22, NHLBI a1111111111 Trans-Omics for Precision Medicine (TOPMed) Consortium, Hematology & Hemostasis, a1111111111 Diabetes, and Structural Variation TOPMed Working Groups¶, Guillaume Lettre6,7³, Vijay a1111111111 G. -

Redalyc.Bases Moleculares De Hemoglobinopatías En Argentina
Acta Bioquímica Clínica Latinoamericana ISSN: 0325-2957 [email protected] Federación Bioquímica de la Provincia de Buenos Aires Argentina Scheps, Karen Gabriela; Varela, Viviana Bases moleculares de hemoglobinopatías en Argentina Acta Bioquímica Clínica Latinoamericana, vol. 51, núm. 3, 2017, pp. 333-342 Federación Bioquímica de la Provincia de Buenos Aires Buenos Aires, Argentina Disponible en: http://www.redalyc.org/articulo.oa?id=53553013008 Cómo citar el artículo Número completo Sistema de Información Científica Más información del artículo Red de Revistas Científicas de América Latina, el Caribe, España y Portugal Página de la revista en redalyc.org Proyecto académico sin fines de lucro, desarrollado bajo la iniciativa de acceso abierto Hematología Reconocimiento a la trayectoria de la Prof. Dra. Nilda Fink Bases moleculares de hemoglobinopatías en Argentina* Molecular basis of hemoglobinopathies in Argentina Bases moleculares de hemoglobinopatias na Argentina ` Karen Gabriela Scheps1,a, Viviana Varela2,a 1 Dra. de la Universidad de Buenos Aires, Área Resumen Biología Molecular. Durante el desarrollo de un individuo se expresan distintas cadenas de globina 2 Dra. de la Universidad de Buenos Aires, Área de tipo y no- , que se combinan en tetrámeros para formar hemoglobina. Los Biología Molecular. Profesora Adjunta de la α α genes que las codifican se organizan en familias. Distintas mutaciones afectan Cátedra de Genética. Facultad de Farmacia y los genes que codifican las cadenas de globina: si provocan alteraciones cua- Bioquímica, Universidad de Buenos Aires. litativas originan cuadros de hemoglobinopatías estructurales, si disminuyen las síntesis de las cadenas de globina, talasemias, y si tienen ambos efectos, a Universidad de Buenos Aires, Cátedra de Ge- hemoglobinopatías talasémicas. -
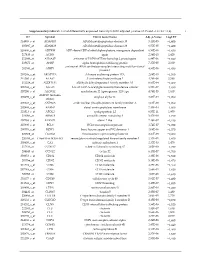
Supplementary Table S1. List of Differentially Expressed
Supplementary table S1. List of differentially expressed transcripts (FDR adjusted p‐value < 0.05 and −1.4 ≤ FC ≥1.4). 1 ID Symbol Entrez Gene Name Adj. p‐Value Log2 FC 214895_s_at ADAM10 ADAM metallopeptidase domain 10 3,11E‐05 −1,400 205997_at ADAM28 ADAM metallopeptidase domain 28 6,57E‐05 −1,400 220606_s_at ADPRM ADP‐ribose/CDP‐alcohol diphosphatase, manganese dependent 6,50E‐06 −1,430 217410_at AGRN agrin 2,34E‐10 1,420 212980_at AHSA2P activator of HSP90 ATPase homolog 2, pseudogene 6,44E‐06 −1,920 219672_at AHSP alpha hemoglobin stabilizing protein 7,27E‐05 2,330 aminoacyl tRNA synthetase complex interacting multifunctional 202541_at AIMP1 4,91E‐06 −1,830 protein 1 210269_s_at AKAP17A A‐kinase anchoring protein 17A 2,64E‐10 −1,560 211560_s_at ALAS2 5ʹ‐aminolevulinate synthase 2 4,28E‐06 3,560 212224_at ALDH1A1 aldehyde dehydrogenase 1 family member A1 8,93E‐04 −1,400 205583_s_at ALG13 ALG13 UDP‐N‐acetylglucosaminyltransferase subunit 9,50E‐07 −1,430 207206_s_at ALOX12 arachidonate 12‐lipoxygenase, 12S type 4,76E‐05 1,630 AMY1C (includes 208498_s_at amylase alpha 1C 3,83E‐05 −1,700 others) 201043_s_at ANP32A acidic nuclear phosphoprotein 32 family member A 5,61E‐09 −1,760 202888_s_at ANPEP alanyl aminopeptidase, membrane 7,40E‐04 −1,600 221013_s_at APOL2 apolipoprotein L2 6,57E‐11 1,600 219094_at ARMC8 armadillo repeat containing 8 3,47E‐08 −1,710 207798_s_at ATXN2L ataxin 2 like 2,16E‐07 −1,410 215990_s_at BCL6 BCL6 transcription repressor 1,74E‐07 −1,700 200776_s_at BZW1 basic leucine zipper and W2 domains 1 1,09E‐06 −1,570 222309_at -

(HBA1 and HBA2) Deletion Duplication with Reflex to Hb
ARUP LABORATORIES | aruplab.com Patient Report 500 Chipeta Way, Salt Lake City, Utah 84108-1221 phone: 801-583-2787, toll free: 800-522-2787 Patient Age/Gender: Unknown Tracy I. George, MD, Chief Medical Officer Specimen Collected: 11-Mar-21 12:41 AlphaThal (HBA1/2) DelDup w/rflx |Received: 11-Mar-21 12:41 Report/Verified: 22-Mar-21 17:02 HbCS FE Procedure Result Units Reference Interval Maternal Contamination Fetal Cells f1 Study Fetal Spec Maternal Contam Study, Whole Blood i1 Maternal Spec Specimen HBA DDCSFE Cultured Amnio HBA DDCSFE See Note f2 i2 Interpretation Result Footnote f1: Maternal Contamination Study Fetal Spec Single fetal genotype present; no maternal cells present. Fetal and maternal samples were tested using STR markers to rule out maternal cell contamination. f2: HBA DDCSFE Interpretation Indication for testing: Prenatal testing for alpha thalassemia. RESULT Two copies of the familial alpha globin deletion were detected. DNA VARIANT Pathogenic Deletion: --SEA; Homozygous Predicted Fetal Genotype: --/-- INTERPRETATION According to information provided to ARUP, both parents of this fetus are carriers of alpha thalassemia and each harbors a heterozygous Southeast Asian (--SEA) deletion. Two copies of the pathogenic --SEA deletion were detected in this fetal sample by deletion/duplication analysis of the alpha globin gene cluster. This result is consistent with the deletion of the HBM, HBA2, HBA1, and HBQ1 globin genes from both chromosomes; thus, this fetus is predicted to be affected with Hb Bart hydrops fetalis syndrome. Evidence for variant classification: The pathogenic --SEA deletion is a common large deletion observed in East Asian populations (HbVar database and references therein). -

32-3942: HBQ1 Recombinant Protein Description
9853 Pacific Heights Blvd. Suite D. San Diego, CA 92121, USA Tel: 858-263-4982 Email: [email protected] 32-3942: HBQ1 Recombinant Protein Alternative Name : Hemoglobin Theta 1,Hemoglobin Theta-1 Chain,Hemoglobin Subunit Theta-1,Theta-1-globin. Description Source : Escherichia Coli. HBQ1 Human Recombinant produced in E.coli is a single, non-glycosylated polypeptide chain containing 165 amino acids (1-142) and having a molecular mass of 17.9kDa.HBQ1 is fused to a 23 amino acid His-tag at N- terminus & purified by proprietary chromatographic techniques. HBQ1 is a member of the Hemoglobin family. Hemoglobin is a 66.7 kDa protein attached to four iron-binding, methenelinked tetrapyrrole rings (heme). The globin part of Hemoglobin contains 2 alpha chains and 2 beta chains organized as couples creating a tetramer. Every one of the 4 globin chains covalently connects with a heme group. The bonds between alpha and beta chains are frailer than between parallel globin chains, thus creating a cleavage plane which is vital for oxygen binding and release. Relaxation of the alpha1-beta2 cleavage plane results in high affinity for oxygen. Product Info Amount : 20 µg Purification : Greater than 90% as determined by SDS-PAGE. The HBQ1 solution contains 20mM Tris-HCl buffer (pH 8.0), 0.15M NaCl, 1mM DTT and 10% Content : glycerol. Store at 4°C if entire vial will be used within 2-4 weeks. Store, frozen at -20°C for longer periods of Storage condition : time. For long term storage it is recommended to add a carrier protein (0.1% HSA or BSA).Avoid multiple freeze-thaw cycles. -

Rayra Pereira Santiago Acidente Vascular...2016.Pdf
0 UNIVERSIDADE FEDERAL DA BAHIA FACULDADE DE MEDICINA FUNDAÇÃO OSWALDO CRUZ CENTRO DE PESQUISAS GONÇALO MONIZ UFBA FIOCRUZ CURSO DE PÓS-GRADUAÇÃO EM PATOLOGIA HUMANA E EXPERIMENTAL DISSERTAÇÃO DE MESTRADO ACIDENTE VASCULAR CEREBRAL NA HEMOGLOBINOPATIA SC (HBB GLU6VAL E GLU6LYS): AVALIAÇÃO DE MARCADORES DE PROGNÓSTICO RAYRA PEREIRA SANTIAGO Salvador – Bahia 2016 1 FUNDAÇÃO OSWALDO CRUZ CENTRO DE PESQUISAS GONÇALO MONIZ Curso de Pós-Graduação em Patologia Humana e Experimental ACIDENTE VASCULAR CEREBRAL NA HEMOGLOBINOPATIA SC (HBB GLU6VAL E GLU6LYS): AVALIAÇÃO DE MARCADORES DE PROGNÓSTICO RAYRA PEREIRA SANTIAGO Orientadora: Profª Drª Marilda de Souza Gonçalves Co-orientadora: Profª Drª Dalila Luciola Zanette Dissertação apresentada ao Curso de Pós-Graduação em Patologia Humana e Experimental para a obtenção do título de Mestre. Salvador – Bahia 2016 2 Ficha Catalográfica elaborada pela Biblioteca do Centro de Pesquisas Gonçalo Moniz / FIOCRUZ - Salvador - Bahia. Santiago, Rayra Pereira S235a Acidente Vascular Cerebral na Hemoglobinopatia SC (HBB glu6val e glu6lys): avaliação de marcadores de prognóstico. / Rayra Pereira Santiago. - 2016. 182 f. : il. ; 30 cm. Orientador: Profª Drª Marilda de Souza Gonçalves, Laboratório de Hematologia, Genética e Biologia Computacional. Dissertação (Mestrado em Patologia) – Fundação Oswaldo Cruz, Centro de Pesquisas Gonçalo Moniz, 2016. 1. Doença SC. 2. Doppler. 3. Acidente vascular cerebral. I. Título. CDU 616.831-005.1 3 “ACIDENTE VASCULAR CEREBRAL NA HEMOGLOBINOPATIA SC (HBB GLU6VAL E GLU6LYS): AVALIAÇÃO DE MARCADORES DE PROGNÓSTICO” RAYRA PEREIRA SANTIAGO FOLHA DE APROVAÇÃO Salvador, 11 de março de 2016 COMISSÃO EXAMINADORA 4 Vamos agradecer a todos, por que nessa vida a gente não faz nada sozinho. Saulo Fernandes 5 Dedico este trabalho aos pacientes com hemoglobinopatia SC, que superam dificuldades todos os dias.