Inline-Supplementary-Material-10.Pdf
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

Tumor Elastography and Its Association with Cell-Free Tumor DNA in the Plasma of Breast Tumor Patients: a Pilot Study
3534 Original Article Tumor elastography and its association with cell-free tumor DNA in the plasma of breast tumor patients: a pilot study Yi Hao1#, Wei Yang2#, Wenyi Zheng2,3#, Xiaona Chen3,4, Hui Wang1,5, Liang Zhao1,5, Jinfeng Xu6,7, Xia Guo4 1Department of Ultrasound, South China Hospital of Shenzhen University, Shenzhen, China; 2Department of Ultrasound, Shenzhen Hospital, Southern Medical University, Shenzhen, China; 3The Third School of Clinical Medicine, Southern Medical University, Guangzhou, China; 4Shenzhen Key Laboratory of Viral Oncology, Center for Clinical Research and Innovation (CCRI), Shenzhen Hospital, Southern Medical University, Shenzhen, China; 5Department of Ultrasound, Affiliated Tumor Hospital of Xinjiang Medical University, Urumqi, China; 6Department of Ultrasound, Shenzhen People’s Hospital (The Second Clinical Medical College, Jinan University, Shenzhen, China; 7The First Affiliated Hospital, Southern University of Science and Technology), Shenzhen, China #These authors contributed equally to this work. Correspondence to: Xia Guo. Shenzhen Key Laboratory of Viral Oncology, Center for Clinical Research and Innovation (CCRI), Shenzhen Hospital, Southern Medical University, Shenzhen 518000, China. Email: [email protected]; Jinfeng Xu. Department of Ultrasound, Shenzhen People’s Hospital (The Second Clinical Medical College, Jinan University, Shenzhen 518020, China; The First Affiliated Hospital, Southern University of Science and Technology), Shenzhen 518020, China. Email: [email protected]. Background: Breast tumor stiffness, which can be objectively and noninvasively evaluated by ultrasound elastography (UE), has been useful for the differentiation of benign and malignant breast lesions and the prediction of clinical outcomes. Liquid biopsy analyses, including cell-free tumor DNA (ctDNA), exhibit great potential for personalized treatment. This study aimed to investigate the correlations between the UE and ctDNA for early breast cancer diagnosis. -

A Computational Approach for Defining a Signature of Β-Cell Golgi Stress in Diabetes Mellitus
Page 1 of 781 Diabetes A Computational Approach for Defining a Signature of β-Cell Golgi Stress in Diabetes Mellitus Robert N. Bone1,6,7, Olufunmilola Oyebamiji2, Sayali Talware2, Sharmila Selvaraj2, Preethi Krishnan3,6, Farooq Syed1,6,7, Huanmei Wu2, Carmella Evans-Molina 1,3,4,5,6,7,8* Departments of 1Pediatrics, 3Medicine, 4Anatomy, Cell Biology & Physiology, 5Biochemistry & Molecular Biology, the 6Center for Diabetes & Metabolic Diseases, and the 7Herman B. Wells Center for Pediatric Research, Indiana University School of Medicine, Indianapolis, IN 46202; 2Department of BioHealth Informatics, Indiana University-Purdue University Indianapolis, Indianapolis, IN, 46202; 8Roudebush VA Medical Center, Indianapolis, IN 46202. *Corresponding Author(s): Carmella Evans-Molina, MD, PhD ([email protected]) Indiana University School of Medicine, 635 Barnhill Drive, MS 2031A, Indianapolis, IN 46202, Telephone: (317) 274-4145, Fax (317) 274-4107 Running Title: Golgi Stress Response in Diabetes Word Count: 4358 Number of Figures: 6 Keywords: Golgi apparatus stress, Islets, β cell, Type 1 diabetes, Type 2 diabetes 1 Diabetes Publish Ahead of Print, published online August 20, 2020 Diabetes Page 2 of 781 ABSTRACT The Golgi apparatus (GA) is an important site of insulin processing and granule maturation, but whether GA organelle dysfunction and GA stress are present in the diabetic β-cell has not been tested. We utilized an informatics-based approach to develop a transcriptional signature of β-cell GA stress using existing RNA sequencing and microarray datasets generated using human islets from donors with diabetes and islets where type 1(T1D) and type 2 diabetes (T2D) had been modeled ex vivo. To narrow our results to GA-specific genes, we applied a filter set of 1,030 genes accepted as GA associated. -

Assembly of an Integrated Human Lung Cell Atlas Reveals That
medRxiv preprint doi: https://doi.org/10.1101/2020.06.02.20120634; this version posted June 4, 2020. The copyright holder for this preprint (which was not certified by peer review) is the author/funder, who has granted medRxiv a license to display the preprint in perpetuity. It is made available under a CC-BY-NC-ND 4.0 International license . Assembly of an integrated human lung cell atlas reveals that SARS-CoV-2 receptor is co-expressed with key elements of the kinin-kallikrein, renin-angiotensin and coagulation systems in alveolar cells Davi Sidarta-Oliveira1,2, Carlos Poblete Jara1,3, Adriano J. Ferruzzi4, Munir S. Skaf4, William H. Velander5, Eliana P. Araujo1,3, Licio A. Velloso1 1Laboratory of Cell Signaling, Obesity and Comorbidities Research Center, University of Campinas, Brazil 2 Physician-Scientist Graduate Program, School of Medical Sciences, University of Campinas, Brazil 3Nursing School, University of Campinas, Brazil 4Institute of Chemistry and Center for Computing in Engineering and Sciences University of Campinas, Brazil 5Department of Chemical and Biomolecular Engineering, University of Nebraska, Lincoln, USA Correspondence: Licio A. Velloso Laboratory of Cell Signaling, Obesity and Comorbidities Research Center, University of Campinas, Campinas, Brazil Address: Rua Carl Von Lineaus s/n, Instituto de Biologia - Bloco Z. Campus Universitário Zeferino Vaz - Barão Geraldo, Campinas - SP, 13083-864 Phone: +55 19 3521-0025 E-mail: [email protected] Abstract SARS-CoV-2, the pathogenic agent of COVID-19, employs angiotensin converting enzyme-2 (ACE2) as its cell entry receptor. Clinical data reveal that in severe COVID- 19, SARS-CoV-2 infects the lung, leading to a frequently lethal triad of respiratory insufficiency, acute cardiovascular failure, and coagulopathy. -

How Relevant Are Bone Marrow-Derived Mast Cells (Bmmcs) As Models for Tissue Mast Cells? a Comparative Transcriptome Analysis of Bmmcs and Peritoneal Mast Cells
cells Article How Relevant Are Bone Marrow-Derived Mast Cells (BMMCs) as Models for Tissue Mast Cells? A Comparative Transcriptome Analysis of BMMCs and Peritoneal Mast Cells 1, 2, 1 1 2,3 Srinivas Akula y , Aida Paivandy y, Zhirong Fu , Michael Thorpe , Gunnar Pejler and Lars Hellman 1,* 1 Department of Cell and Molecular Biology, Uppsala University, The Biomedical Center, Box 596, SE-751 24 Uppsala, Sweden; [email protected] (S.A.); [email protected] (Z.F.); [email protected] (M.T.) 2 Department of Medical Biochemistry and Microbiology, Uppsala University, The Biomedical Center, Box 589, SE-751 23 Uppsala, Sweden; [email protected] (A.P.); [email protected] (G.P.) 3 Department of Anatomy, Physiology and Biochemistry, Swedish University of Agricultural Sciences, Box 7011, SE-75007 Uppsala, Sweden * Correspondence: [email protected]; Tel.: +46-(0)18-471-4532; Fax: +46-(0)18-471-4862 These authors contributed equally to this work. y Received: 29 July 2020; Accepted: 16 September 2020; Published: 17 September 2020 Abstract: Bone marrow-derived mast cells (BMMCs) are often used as a model system for studies of the role of MCs in health and disease. These cells are relatively easy to obtain from total bone marrow cells by culturing under the influence of IL-3 or stem cell factor (SCF). After 3 to 4 weeks in culture, a nearly homogenous cell population of toluidine blue-positive cells are often obtained. However, the question is how relevant equivalents these cells are to normal tissue MCs. By comparing the total transcriptome of purified peritoneal MCs with BMMCs, here we obtained a comparative view of these cells. -

1 No. Affymetrix ID Gene Symbol Genedescription Gotermsbp Q Value 1. 209351 at KRT14 Keratin 14 Structural Constituent of Cyto
1 Affymetrix Gene Q No. GeneDescription GOTermsBP ID Symbol value structural constituent of cytoskeleton, intermediate 1. 209351_at KRT14 keratin 14 filament, epidermis development <0.01 biological process unknown, S100 calcium binding calcium ion binding, cellular 2. 204268_at S100A2 protein A2 component unknown <0.01 regulation of progression through cell cycle, extracellular space, cytoplasm, cell proliferation, protein kinase C inhibitor activity, protein domain specific 3. 33323_r_at SFN stratifin/14-3-3σ binding <0.01 regulation of progression through cell cycle, extracellular space, cytoplasm, cell proliferation, protein kinase C inhibitor activity, protein domain specific 4. 33322_i_at SFN stratifin/14-3-3σ binding <0.01 structural constituent of cytoskeleton, intermediate 5. 201820_at KRT5 keratin 5 filament, epidermis development <0.01 structural constituent of cytoskeleton, intermediate 6. 209125_at KRT6A keratin 6A filament, ectoderm development <0.01 regulation of progression through cell cycle, extracellular space, cytoplasm, cell proliferation, protein kinase C inhibitor activity, protein domain specific 7. 209260_at SFN stratifin/14-3-3σ binding <0.01 structural constituent of cytoskeleton, intermediate 8. 213680_at KRT6B keratin 6B filament, ectoderm development <0.01 receptor activity, cytosol, integral to plasma membrane, cell surface receptor linked signal transduction, sensory perception, tumor-associated calcium visual perception, cell 9. 202286_s_at TACSTD2 signal transducer 2 proliferation, membrane <0.01 structural constituent of cytoskeleton, cytoskeleton, intermediate filament, cell-cell adherens junction, epidermis 10. 200606_at DSP desmoplakin development <0.01 lectin, galactoside- sugar binding, extracellular binding, soluble, 7 space, nucleus, apoptosis, 11. 206400_at LGALS7 (galectin 7) heterophilic cell adhesion <0.01 2 S100 calcium binding calcium ion binding, epidermis 12. 205916_at S100A7 protein A7 (psoriasin 1) development <0.01 S100 calcium binding protein A8 (calgranulin calcium ion binding, extracellular 13. -

Development and Validation of a Protein-Based Risk Score for Cardiovascular Outcomes Among Patients with Stable Coronary Heart Disease
Supplementary Online Content Ganz P, Heidecker B, Hveem K, et al. Development and validation of a protein-based risk score for cardiovascular outcomes among patients with stable coronary heart disease. JAMA. doi: 10.1001/jama.2016.5951 eTable 1. List of 1130 Proteins Measured by Somalogic’s Modified Aptamer-Based Proteomic Assay eTable 2. Coefficients for Weibull Recalibration Model Applied to 9-Protein Model eFigure 1. Median Protein Levels in Derivation and Validation Cohort eTable 3. Coefficients for the Recalibration Model Applied to Refit Framingham eFigure 2. Calibration Plots for the Refit Framingham Model eTable 4. List of 200 Proteins Associated With the Risk of MI, Stroke, Heart Failure, and Death eFigure 3. Hazard Ratios of Lasso Selected Proteins for Primary End Point of MI, Stroke, Heart Failure, and Death eFigure 4. 9-Protein Prognostic Model Hazard Ratios Adjusted for Framingham Variables eFigure 5. 9-Protein Risk Scores by Event Type This supplementary material has been provided by the authors to give readers additional information about their work. Downloaded From: https://jamanetwork.com/ on 10/02/2021 Supplemental Material Table of Contents 1 Study Design and Data Processing ......................................................................................................... 3 2 Table of 1130 Proteins Measured .......................................................................................................... 4 3 Variable Selection and Statistical Modeling ........................................................................................ -
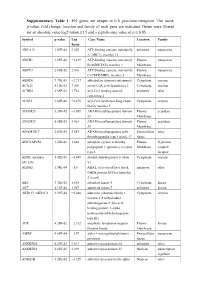
Supplementary Table 1
Supplementary Table 1. 492 genes are unique to 0 h post-heat timepoint. The name, p-value, fold change, location and family of each gene are indicated. Genes were filtered for an absolute value log2 ration 1.5 and a significance value of p ≤ 0.05. Symbol p-value Log Gene Name Location Family Ratio ABCA13 1.87E-02 3.292 ATP-binding cassette, sub-family unknown transporter A (ABC1), member 13 ABCB1 1.93E-02 −1.819 ATP-binding cassette, sub-family Plasma transporter B (MDR/TAP), member 1 Membrane ABCC3 2.83E-02 2.016 ATP-binding cassette, sub-family Plasma transporter C (CFTR/MRP), member 3 Membrane ABHD6 7.79E-03 −2.717 abhydrolase domain containing 6 Cytoplasm enzyme ACAT1 4.10E-02 3.009 acetyl-CoA acetyltransferase 1 Cytoplasm enzyme ACBD4 2.66E-03 1.722 acyl-CoA binding domain unknown other containing 4 ACSL5 1.86E-02 −2.876 acyl-CoA synthetase long-chain Cytoplasm enzyme family member 5 ADAM23 3.33E-02 −3.008 ADAM metallopeptidase domain Plasma peptidase 23 Membrane ADAM29 5.58E-03 3.463 ADAM metallopeptidase domain Plasma peptidase 29 Membrane ADAMTS17 2.67E-04 3.051 ADAM metallopeptidase with Extracellular other thrombospondin type 1 motif, 17 Space ADCYAP1R1 1.20E-02 1.848 adenylate cyclase activating Plasma G-protein polypeptide 1 (pituitary) receptor Membrane coupled type I receptor ADH6 (includes 4.02E-02 −1.845 alcohol dehydrogenase 6 (class Cytoplasm enzyme EG:130) V) AHSA2 1.54E-04 −1.6 AHA1, activator of heat shock unknown other 90kDa protein ATPase homolog 2 (yeast) AK5 3.32E-02 1.658 adenylate kinase 5 Cytoplasm kinase AK7 -

Asthma Experimental Involved in the Development of Mouse Mast Cell Protease-6 and MHC
Mouse Mast Cell Protease-6 and MHC Are Involved in the Development of Experimental Asthma This information is current as Yue Cui, Joakim S. Dahlin, Ricardo Feinstein, Lora G. of September 28, 2021. Bankova, Wei Xing, Kichul Shin, Michael F. Gurish and Jenny Hallgren J Immunol 2014; 193:4783-4789; Prepublished online 15 October 2014; doi: 10.4049/jimmunol.1302947 Downloaded from http://www.jimmunol.org/content/193/10/4783 References This article cites 33 articles, 16 of which you can access for free at: http://www.jimmunol.org/content/193/10/4783.full#ref-list-1 http://www.jimmunol.org/ Why The JI? Submit online. • Rapid Reviews! 30 days* from submission to initial decision • No Triage! Every submission reviewed by practicing scientists by guest on September 28, 2021 • Fast Publication! 4 weeks from acceptance to publication *average Subscription Information about subscribing to The Journal of Immunology is online at: http://jimmunol.org/subscription Permissions Submit copyright permission requests at: http://www.aai.org/About/Publications/JI/copyright.html Email Alerts Receive free email-alerts when new articles cite this article. Sign up at: http://jimmunol.org/alerts The Journal of Immunology is published twice each month by The American Association of Immunologists, Inc., 1451 Rockville Pike, Suite 650, Rockville, MD 20852 Copyright © 2014 by The American Association of Immunologists, Inc. All rights reserved. Print ISSN: 0022-1767 Online ISSN: 1550-6606. The Journal of Immunology Mouse Mast Cell Protease-6 and MHC Are Involved in the Development of Experimental Asthma Yue Cui,*,1 Joakim S. Dahlin,*,1 Ricardo Feinstein,† Lora G. -

Mast Cell Secretory Granules: Armed for Battle
REVIEWS Mast cell secretory granules: armed for battle Sara Wernersson and Gunnar Pejler Abstract | Mast cells are important effector cells of the immune system and recent studies show that they have immunomodulatory roles in diverse processes in both health and disease. Mast cells are distinguished by their high content of electron-dense secretory granules, which are filled with large amounts of preformed and pre-activated immunomodulatory compounds. When appropriately activated, mast cells undergo degranulation, a process by which these preformed granule compounds are rapidly released into the surroundings. In many cases, the effects that mast cells have on an immune response are closely associated with the biological actions of the granule compounds that they release, as exemplified by the recent studies showing that mast cell granule proteases account for many of the protective and detrimental effects of mast cells in various inflammatory settings. In this Review, we discuss the current knowledge of mast cell secretory granules. Tr y p ta s e s Mast cells are haematopoietic cells that arise from pluri- The secretory granules of mast cells are filled with a 1–3 Serine proteases that have potent precursors of the bone marrow . After egression large panel of preformed compounds (TABLE 1). When trypsin-like cleavage from the bone marrow, mast cell progenitors circu- mast cells are activated to degranulate, these com- specificities — that is, they late in the blood before they enter various tissues and pounds are released into the extracellular environment cleave peptide bonds on the develop into mature mast cells under the influence of and can have a marked effect on any physiological or carboxy-terminal side of arginine or lysine residues. -

Download Guide
Understanding your results This test is used to determine alpha tryptase copy number. Alpha tryptase is encoded by the TPSAB1 gene which is on chromosome 16. The primary gene products of this gene are β-tryptase, which can be produced by TPSAB1 and TPSB2, and α-tryptase, which is only produced by the TPSAB1 gene. Individuals who inherited one copy of α-tryptase–encoding sequence on both have normal serum tryptase levels. This test measures both α- and β-tryptase. A normal result is any combination of α- and β-tryptase that adds up to 4. A positive result – which means there is a duplication or triplication of α-trypase – is a combination of α- and β-tryptase that is 5 or higher. An explanation of the results are included below, however there may be other copy number changes in these genes that have not yet been described. In addition, the assay cannot distinguish between the possible 4,2 genotypes without testing of additional family members. NORMAL RESULT: Alpha-tryptase copy number 2; beta-tryptase copy number 2 TPSB2 TPSAB1 β α β α NORMAL RESULT: Alpha-tryptase copy number 1; beta-tryptase copy number 3 TPSB2 TPSAB1 β β β α 1445 N. Loop W., Suite 820, Houston, TX 77008 (713)868-1438 CLIA#45D1102202 CAP#7212851 NORMAL RESULT: Alpha-tryptase copy number 0; beta-tryptase copy number 4 TPSB2 TPSAB1 β β β β POSITIVE RESULT: Alpha-tryptase copy number 2; beta-tryptase copy number 3 TPSB2 TPSAB1 β α α β β POSITIVE RESULT: Alpha-tryptase copy number 3; beta-tryptase copy number 2 TPSB2 TPSAB1 β α α β α 1445 N. -

A Genomic Analysis of Rat Proteases and Protease Inhibitors
A genomic analysis of rat proteases and protease inhibitors Xose S. Puente and Carlos López-Otín Departamento de Bioquímica y Biología Molecular, Facultad de Medicina, Instituto Universitario de Oncología, Universidad de Oviedo, 33006-Oviedo, Spain Send correspondence to: Carlos López-Otín Departamento de Bioquímica y Biología Molecular Facultad de Medicina, Universidad de Oviedo 33006 Oviedo-SPAIN Tel. 34-985-104201; Fax: 34-985-103564 E-mail: [email protected] Proteases perform fundamental roles in multiple biological processes and are associated with a growing number of pathological conditions that involve abnormal or deficient functions of these enzymes. The availability of the rat genome sequence has opened the possibility to perform a global analysis of the complete protease repertoire or degradome of this model organism. The rat degradome consists of at least 626 proteases and homologs, which are distributed into five catalytic classes: 24 aspartic, 160 cysteine, 192 metallo, 221 serine, and 29 threonine proteases. Overall, this distribution is similar to that of the mouse degradome, but significatively more complex than that corresponding to the human degradome composed of 561 proteases and homologs. This increased complexity of the rat protease complement mainly derives from the expansion of several gene families including placental cathepsins, testases, kallikreins and hematopoietic serine proteases, involved in reproductive or immunological functions. These protease families have also evolved differently in the rat and mouse genomes and may contribute to explain some functional differences between these two closely related species. Likewise, genomic analysis of rat protease inhibitors has shown some differences with the mouse protease inhibitor complement and the marked expansion of families of cysteine and serine protease inhibitors in rat and mouse with respect to human. -

Hereditary Alpha Tryptasemia, Mastocytosis and Beyond
International Journal of Molecular Sciences Review Genetic Regulation of Tryptase Production and Clinical Impact: Hereditary Alpha Tryptasemia, Mastocytosis and Beyond Bettina Sprinzl 1,2, Georg Greiner 3,4,5 , Goekhan Uyanik 1,2,6, Michel Arock 7,8 , Torsten Haferlach 9, Wolfgang R. Sperr 4,10, Peter Valent 4,10 and Gregor Hoermann 4,9,* 1 Ludwig Boltzmann Institute for Hematology and Oncology at the Hanusch Hospital, Center for Medical Genetics, Hanusch Hospital, 1140 Vienna, Austria; [email protected] (B.S.); [email protected] (G.U.) 2 Center for Medical Genetics, Hanusch Hospital, 1140 Vienna, Austria 3 Department of Laboratory Medicine, Medical University of Vienna, 1090 Vienna, Austria; [email protected] 4 Ludwig Boltzmann Institute for Hematology and Oncology, Medical University of Vienna, 1090 Vienna, Austria; [email protected] (W.R.S.); [email protected] (P.V.) 5 Ihr Labor, Medical Diagnostic Laboratories, 1220 Vienna, Austria 6 Medical School, Sigmund Freud Private University, 1020 Vienna, Austria 7 Department of Hematology, APHP, Pitié-Salpêtrière-Charles Foix University Hospital and Sorbonne University, 75013 Paris, France; [email protected] 8 Centre de Recherche des Cordeliers, INSERM, Sorbonne University, Cell Death and Drug Resistance in Hematological Disorders Team, 75006 Paris, France 9 MLL Munich Leukemia Laboratory, 81377 Munich, Germany; [email protected] 10 Department of Internal Medicine I, Division of Hematology and Hemostaseology, Medical University of Vienna, 1090 Vienna, Austria * Correspondence: [email protected]; Tel.: +49-89-99017-315 Citation: Sprinzl, B.; Greiner, G.; Uyanik, G.; Arock, M.; Haferlach, T.; Abstract: Tryptase is a serine protease that is predominantly produced by tissue mast cells (MCs) and Sperr, W.R.; Valent, P.; Hoermann, G.