Supplemental Data Tuberous Sclerosis Complex-Associated CNS
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

EGFR and Mtor As Therapeutic Targets in Glioblastoma
www.oncotarget.com Oncotarget, 2019, Vol. 10, (No. 46), pp: 4721-4723 Editorial EGFR and mTOR as therapeutic targets in glioblastoma Michael W. Ronellenfitsch, Anna-Luisa Luger and Joachim P. Steinbach The quest for new and improved therapies for mammalian target of rapamycin complex 1 (mTORC1) glioblastoma (GB) has been mostly unsuccessful in signaling were found in the majority of GBs [3]. more than a decade despite significant efforts. The few Therefore, many hopes have rested on targeted therapies. exceptions include the optimization of classical alkylating However, the results from clinical trials have been largely chemotherapy by including lomustine in the first line disappointing [4]. Nevertheless, unplanned retrospective regimen for GB with a methylated MGMT promoter and subgroup analyses of the patient cohorts of negative tumor treating fields [1, 2]. The GB signaling network has clinical trials indicated that dysregulation or activation been well-characterized and genetic alterations resulting of signaling could be a predictive factor for susceptibility in activation of receptor tyrosine kinases and especially to pathway inhibition: Tumors with enhanced levels of epidermal growth factor receptor (EGFR) and downstream mTORC1 activation markers, including phosphorylated Figure 1: Scheme of EGFR signaling and DDIT4-mediated adaptive processes. Conditions of the glioblastoma microenvironment including hypoxia, alkylating therapy or irradiation trigger induction of DDIT4 which activates TSC1/2 to inhibit mTORC1 and can counteract epidermal growth factor receptor (EGFR)-mediated TSC1/2 inhibition. Inhibition of mTORC1 ultimately induces adaptive processes to cope with external stressors. www.oncotarget.com 4721 Oncotarget ribosomal protein S6 and phosphorylated mTOR itself, of effects. Depending on the half life and pharmacokinetics appeared to respond to pathway inhibition by the EGFR of the drugs, stepwise treatment algorithms could be an antibody nimotuzumab or the mTORC1 inhibitor option to prevent antagonistic effects. -
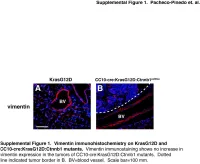
Supplemental Figure 1. Vimentin
Double mutant specific genes Transcript gene_assignment Gene Symbol RefSeq FDR Fold- FDR Fold- FDR Fold- ID (single vs. Change (double Change (double Change wt) (single vs. wt) (double vs. single) (double vs. wt) vs. wt) vs. single) 10485013 BC085239 // 1110051M20Rik // RIKEN cDNA 1110051M20 gene // 2 E1 // 228356 /// NM 1110051M20Ri BC085239 0.164013 -1.38517 0.0345128 -2.24228 0.154535 -1.61877 k 10358717 NM_197990 // 1700025G04Rik // RIKEN cDNA 1700025G04 gene // 1 G2 // 69399 /// BC 1700025G04Rik NM_197990 0.142593 -1.37878 0.0212926 -3.13385 0.093068 -2.27291 10358713 NM_197990 // 1700025G04Rik // RIKEN cDNA 1700025G04 gene // 1 G2 // 69399 1700025G04Rik NM_197990 0.0655213 -1.71563 0.0222468 -2.32498 0.166843 -1.35517 10481312 NM_027283 // 1700026L06Rik // RIKEN cDNA 1700026L06 gene // 2 A3 // 69987 /// EN 1700026L06Rik NM_027283 0.0503754 -1.46385 0.0140999 -2.19537 0.0825609 -1.49972 10351465 BC150846 // 1700084C01Rik // RIKEN cDNA 1700084C01 gene // 1 H3 // 78465 /// NM_ 1700084C01Rik BC150846 0.107391 -1.5916 0.0385418 -2.05801 0.295457 -1.29305 10569654 AK007416 // 1810010D01Rik // RIKEN cDNA 1810010D01 gene // 7 F5 // 381935 /// XR 1810010D01Rik AK007416 0.145576 1.69432 0.0476957 2.51662 0.288571 1.48533 10508883 NM_001083916 // 1810019J16Rik // RIKEN cDNA 1810019J16 gene // 4 D2.3 // 69073 / 1810019J16Rik NM_001083916 0.0533206 1.57139 0.0145433 2.56417 0.0836674 1.63179 10585282 ENSMUST00000050829 // 2010007H06Rik // RIKEN cDNA 2010007H06 gene // --- // 6984 2010007H06Rik ENSMUST00000050829 0.129914 -1.71998 0.0434862 -2.51672 -

Exosomal Lncrna DOCK9-AS2 Derived from Cancer Stem Cell-Like
Dai et al. Cell Death and Disease (2020) 11:743 https://doi.org/10.1038/s41419-020-02827-w Cell Death & Disease ARTICLE Open Access Exosomal lncRNA DOCK9-AS2 derived from cancer stem cell-like cells activated Wnt/β-catenin pathway to aggravate stemness, proliferation, migration, and invasion in papillary thyroid carcinoma Wencheng Dai1, Xiaoxia Jin2,LiangHan1, Haijing Huang3,ZhenhuaJi1, Xinjiang Xu1, Mingming Tang1,BinJiang1 and Weixian Chen1 Abstract Exosomal long non-coding RNAs (lncRNAs) are crucial factors that mediate the extracellular communication in tumor microenvironment. DOCK9 antisense RNA2 (DOCK9-AS2) is an exosomal lncRNA which has not been investigated in papillary thyroid carcinoma (PTC). Based on the result of differentially expressed lncRNAs in PTC via bioinformatics databases, we discovered that DOCK9-AS2 was upregulated in PTC, and presented elevation in plasma exosomes of PTC patients. Functionally, DOCK9-AS2 knockdown reduced proliferation, migration, invasion, epithelial-to- mesenchymal (EMT) and stemness in PTC cells. PTC-CSCs transmitted exosomal DOCK9-AS2 to improve stemness of PTC cells. Mechanistically, DOCK9-AS2 interacted with SP1 to induce catenin beta 1 (CTNNB1) transcription and 1234567890():,; 1234567890():,; 1234567890():,; 1234567890():,; sponged microRNA-1972 (miR-1972) to upregulate CTNNB1, thereby activating Wnt/β-catenin pathway in PTC cells. In conclusion, PTC-CSCs-derived exosomal lncRNA DOCK9-AS2 activated Wnt/β-catenin pathway to aggravate PTC progression, indicating that DOCK9-AS2 was a potential target for therapies in PTC. Introduction Therefore, more efforts are required for the improvement of Papillary thyroid cancer (PTC) takes up around 80% of targeted therapy and diagnosis in PTC. thyroid cancer (TC) cases1. Treatment outcome of PTC is Long non-coding RNAs (lncRNAs) are known as tran- generally satisfactory, and with appropriate treatment, over scripts without protein-coding ability and consist over 200 95% of PTC patients can survive longer than 5 years2. -

Implications in Parkinson's Disease
Journal of Clinical Medicine Review Lysosomal Ceramide Metabolism Disorders: Implications in Parkinson’s Disease Silvia Paciotti 1,2 , Elisabetta Albi 3 , Lucilla Parnetti 1 and Tommaso Beccari 3,* 1 Laboratory of Clinical Neurochemistry, Department of Medicine, University of Perugia, Sant’Andrea delle Fratte, 06132 Perugia, Italy; [email protected] (S.P.); [email protected] (L.P.) 2 Section of Physiology and Biochemistry, Department of Experimental Medicine, University of Perugia, Sant’Andrea delle Fratte, 06132 Perugia, Italy 3 Department of Pharmaceutical Sciences, University of Perugia, Via Fabretti, 06123 Perugia, Italy; [email protected] * Correspondence: [email protected] Received: 29 January 2020; Accepted: 20 February 2020; Published: 21 February 2020 Abstract: Ceramides are a family of bioactive lipids belonging to the class of sphingolipids. Sphingolipidoses are a group of inherited genetic diseases characterized by the unmetabolized sphingolipids and the consequent reduction of ceramide pool in lysosomes. Sphingolipidoses include several disorders as Sandhoff disease, Fabry disease, Gaucher disease, metachromatic leukodystrophy, Krabbe disease, Niemann Pick disease, Farber disease, and GM2 gangliosidosis. In sphingolipidosis, lysosomal lipid storage occurs in both the central nervous system and visceral tissues, and central nervous system pathology is a common hallmark for all of them. Parkinson’s disease, the most common neurodegenerative movement disorder, is characterized by the accumulation and aggregation of misfolded α-synuclein that seem associated to some lysosomal disorders, in particular Gaucher disease. This review provides evidence into the role of ceramide metabolism in the pathophysiology of lysosomes, highlighting the more recent findings on its involvement in Parkinson’s disease. Keywords: ceramide metabolism; Parkinson’s disease; α-synuclein; GBA; GLA; HEX A-B; GALC; ASAH1; SMPD1; ARSA * Correspondence [email protected] 1. -

Regulation of Cdc42 and Its Effectors in Epithelial Morphogenesis Franck Pichaud1,2,*, Rhian F
© 2019. Published by The Company of Biologists Ltd | Journal of Cell Science (2019) 132, jcs217869. doi:10.1242/jcs.217869 REVIEW SUBJECT COLLECTION: ADHESION Regulation of Cdc42 and its effectors in epithelial morphogenesis Franck Pichaud1,2,*, Rhian F. Walther1 and Francisca Nunes de Almeida1 ABSTRACT An overview of Cdc42 Cdc42 – a member of the small Rho GTPase family – regulates cell Cdc42 was discovered in yeast and belongs to a large family of small – polarity across organisms from yeast to humans. It is an essential (20 30 kDa) GTP-binding proteins (Adams et al., 1990; Johnson regulator of polarized morphogenesis in epithelial cells, through and Pringle, 1990). It is part of the Ras-homologous Rho subfamily coordination of apical membrane morphogenesis, lumen formation and of GTPases, of which there are 20 members in humans, including junction maturation. In parallel, work in yeast and Caenorhabditis elegans the RhoA and Rac GTPases, (Hall, 2012). Rho, Rac and Cdc42 has provided important clues as to how this molecular switch can homologues are found in all eukaryotes, except for plants, which do generate and regulate polarity through localized activation or inhibition, not have a clear homologue for Cdc42. Together, the function of and cytoskeleton regulation. Recent studies have revealed how Rho GTPases influences most, if not all, cellular processes. important and complex these regulations can be during epithelial In the early 1990s, seminal work from Alan Hall and his morphogenesis. This complexity is mirrored by the fact that Cdc42 can collaborators identified Rho, Rac and Cdc42 as main regulators of exert its function through many effector proteins. -

The Atypical Guanine-Nucleotide Exchange Factor, Dock7, Negatively Regulates Schwann Cell Differentiation and Myelination
The Journal of Neuroscience, August 31, 2011 • 31(35):12579–12592 • 12579 Cellular/Molecular The Atypical Guanine-Nucleotide Exchange Factor, Dock7, Negatively Regulates Schwann Cell Differentiation and Myelination Junji Yamauchi,1,3,5 Yuki Miyamoto,1 Hajime Hamasaki,1,3 Atsushi Sanbe,1 Shinji Kusakawa,1 Akane Nakamura,2 Hideki Tsumura,2 Masahiro Maeda,4 Noriko Nemoto,6 Katsumasa Kawahara,5 Tomohiro Torii,1 and Akito Tanoue1 1Department of Pharmacology and 2Laboratory Animal Resource Facility, National Research Institute for Child Health and Development, Setagaya, Tokyo 157-8535, Japan, 3Department of Biological Sciences, Tokyo Institute of Technology, Midori, Yokohama 226-8501, Japan, 4IBL, Ltd., Fujioka, Gumma 375-0005, Japan, and 5Department of Physiology and 6Bioimaging Research Center, Kitasato University School of Medicine, Sagamihara, Kanagawa 252-0374, Japan In development of the peripheral nervous system, Schwann cells proliferate, migrate, and ultimately differentiate to form myelin sheath. In all of the myelination stages, Schwann cells continuously undergo morphological changes; however, little is known about their underlying molecular mechanisms. We previously cloned the dock7 gene encoding the atypical Rho family guanine-nucleotide exchange factor (GEF) and reported the positive role of Dock7, the target Rho GTPases Rac/Cdc42, and the downstream c-Jun N-terminal kinase in Schwann cell migration (Yamauchi et al., 2008). We investigated the role of Dock7 in Schwann cell differentiation and myelination. Knockdown of Dock7 by the specific small interfering (si)RNA in primary Schwann cells promotes dibutyryl cAMP-induced morpholog- ical differentiation, indicating the negative role of Dock7 in Schwann cell differentiation. It also results in a shorter duration of activation of Rac/Cdc42 and JNK, which is the negative regulator of myelination, and the earlier activation of Rho and Rho-kinase, which is the positive regulator of myelination. -

A Computational Approach for Defining a Signature of Β-Cell Golgi Stress in Diabetes Mellitus
Page 1 of 781 Diabetes A Computational Approach for Defining a Signature of β-Cell Golgi Stress in Diabetes Mellitus Robert N. Bone1,6,7, Olufunmilola Oyebamiji2, Sayali Talware2, Sharmila Selvaraj2, Preethi Krishnan3,6, Farooq Syed1,6,7, Huanmei Wu2, Carmella Evans-Molina 1,3,4,5,6,7,8* Departments of 1Pediatrics, 3Medicine, 4Anatomy, Cell Biology & Physiology, 5Biochemistry & Molecular Biology, the 6Center for Diabetes & Metabolic Diseases, and the 7Herman B. Wells Center for Pediatric Research, Indiana University School of Medicine, Indianapolis, IN 46202; 2Department of BioHealth Informatics, Indiana University-Purdue University Indianapolis, Indianapolis, IN, 46202; 8Roudebush VA Medical Center, Indianapolis, IN 46202. *Corresponding Author(s): Carmella Evans-Molina, MD, PhD ([email protected]) Indiana University School of Medicine, 635 Barnhill Drive, MS 2031A, Indianapolis, IN 46202, Telephone: (317) 274-4145, Fax (317) 274-4107 Running Title: Golgi Stress Response in Diabetes Word Count: 4358 Number of Figures: 6 Keywords: Golgi apparatus stress, Islets, β cell, Type 1 diabetes, Type 2 diabetes 1 Diabetes Publish Ahead of Print, published online August 20, 2020 Diabetes Page 2 of 781 ABSTRACT The Golgi apparatus (GA) is an important site of insulin processing and granule maturation, but whether GA organelle dysfunction and GA stress are present in the diabetic β-cell has not been tested. We utilized an informatics-based approach to develop a transcriptional signature of β-cell GA stress using existing RNA sequencing and microarray datasets generated using human islets from donors with diabetes and islets where type 1(T1D) and type 2 diabetes (T2D) had been modeled ex vivo. To narrow our results to GA-specific genes, we applied a filter set of 1,030 genes accepted as GA associated. -

Understanding the Molecular Pathobiology of Acid Ceramidase Deficiency
Understanding the Molecular Pathobiology of Acid Ceramidase Deficiency By Fabian Yu A thesis submitted in conformity with the requirements for the degree of Doctor of Philosophy Institute of Medical Science University of Toronto © Copyright by Fabian PS Yu 2018 Understanding the Molecular Pathobiology of Acid Ceramidase Deficiency Fabian Yu Doctor of Philosophy Institute of Medical Science University of Toronto 2018 Abstract Farber disease (FD) is a devastating Lysosomal Storage Disorder (LSD) caused by mutations in ASAH1, resulting in acid ceramidase (ACDase) deficiency. ACDase deficiency manifests along a broad spectrum but in its classical form patients die during early childhood. Due to the scarcity of cases FD has largely been understudied. To circumvent this, our lab previously generated a mouse model that recapitulates FD. In some case reports, patients have shown signs of visceral involvement, retinopathy and respiratory distress that may lead to death. Beyond superficial descriptions in case reports, there have been no in-depth studies performed to address these conditions. To improve the understanding of FD and gain insights for evaluating future therapies, we performed comprehensive studies on the ACDase deficient mouse. In the visual system, we reported presence of progressive uveitis. Further tests revealed cellular infiltration, lipid buildup and extensive retinal pathology. Mice developed retinal dysplasia, impaired retinal response and decreased visual acuity. Within the pulmonary system, lung function tests revealed a decrease in lung compliance. Mice developed chronic lung injury that was contributed by cellular recruitment, and vascular leakage. Additionally, we report impairment to lipid homeostasis in the lungs. ii To understand the liver involvement in FD, we characterized the pathology and performed transcriptome analysis to identify gene and pathway changes. -

Effects of Rapamycin on Social Interaction Deficits and Gene
Kotajima-Murakami et al. Molecular Brain (2019) 12:3 https://doi.org/10.1186/s13041-018-0423-2 RESEARCH Open Access Effects of rapamycin on social interaction deficits and gene expression in mice exposed to valproic acid in utero Hiroko Kotajima-Murakami1,2, Toshiyuki Kobayashi3, Hirofumi Kashii1,4, Atsushi Sato1,5, Yoko Hagino1, Miho Tanaka1,6, Yasumasa Nishito7, Yukio Takamatsu7, Shigeo Uchino1,2 and Kazutaka Ikeda1* Abstract The mammalian target of rapamycin (mTOR) signaling pathway plays a crucial role in cell metabolism, growth, and proliferation. The overactivation of mTOR has been implicated in the pathogenesis of syndromic autism spectrum disorder (ASD), such as tuberous sclerosis complex (TSC). Treatment with the mTOR inhibitor rapamycin improved social interaction deficits in mouse models of TSC. Prenatal exposure to valproic acid (VPA) increases the incidence of ASD. Rodent pups that are exposed to VPA in utero have been used as an animal model of ASD. Activation of the mTOR signaling pathway was recently observed in rodents that were exposed to VPA in utero, and rapamycin ameliorated social interaction deficits. The present study investigated the effect of rapamycin on social interaction deficits in both adolescence and adulthood, and gene expressions in mice that were exposed to VPA in utero. We subcutaneously injected 600 mg/kg VPA in pregnant mice on gestational day 12.5 and used the pups as a model of ASD. The pups were intraperitoneally injected with rapamycin or an equal volume of vehicle once daily for 2 consecutive days. The social interaction test was conducted in the offspring after the last rapamycin administration at 5–6 weeks of ages (adolescence) or 10–11 weeks of age (adulthood). -

The Role of High Density Lipoprotein Compositional and Functional Heterogeneity in Metabolic Disease
The role of high density lipoprotein compositional and functional heterogeneity in metabolic disease By Scott M. Gordon B.S. State University of New York College at Brockport October, 2012 A Dissertation Presented to the Faculty of The University of Cincinnati College of Medicine in partial fulfillment of the requirements for the Degree of Doctor of Philosophy from the Pathobiology and Molecular Medicine graduate program W. Sean Davidson Ph.D. (Chair) David Askew Ph.D. Professor and Thesis Chair Professor Department of Pathology Department of Pathology University of Cincinnati University of Cincinnati Francis McCormack M.D. Gangani Silva Ph.D. Professor Assistant Professor Department of Pathology Department of Pathology University of Cincinnati University of Cincinnati Jason Lu Ph.D. Assistant Professor Division of Bioinformatics Cincinnati Children’s Hospital i Abstract High density lipoproteins (HDL) are complexes of phospholipid, cholesterol and protein that circulate in the blood. Epidemiological studies have demonstrated a strong inverse correlation between plasma levels of HDL associated cholesterol (HDL-C) and the incidence of cardiovascular disease (CVD). Clinically, HDL-C is often measured and used in combination with low density lipoprotein cholesterol (LDL-C) to assess overall cardiovascular health. HDL have been shown to possess a wide variety of functional attributes which likely contribute to this protection including anti-inflammatory and anti- oxidative properties and the ability to remove excess cholesterol from peripheral tissues and deliver it to the liver for excretion, a process known as reverse cholesterol transport. This functional diversity might be explained by the complexity of HDL composition. Recent studies have taken advantage of advances in mass spectrometry technologies to characterize the proteome of total HDL finding that over 50 different proteins can associate with these particles. -

A Rac/Cdc42 Exchange Factor Complex Promotes Formation of Lateral filopodia and Blood Vessel Lumen Morphogenesis
ARTICLE Received 1 Oct 2014 | Accepted 26 Apr 2015 | Published 1 Jul 2015 DOI: 10.1038/ncomms8286 OPEN A Rac/Cdc42 exchange factor complex promotes formation of lateral filopodia and blood vessel lumen morphogenesis Sabu Abraham1,w,*, Margherita Scarcia2,w,*, Richard D. Bagshaw3,w,*, Kathryn McMahon2,w, Gary Grant2, Tracey Harvey2,w, Maggie Yeo1, Filomena O.G. Esteves2, Helene H. Thygesen2,w, Pamela F. Jones4, Valerie Speirs2, Andrew M. Hanby2, Peter J. Selby2, Mihaela Lorger2, T. Neil Dear4,w, Tony Pawson3,z, Christopher J. Marshall1 & Georgia Mavria2 During angiogenesis, Rho-GTPases influence endothelial cell migration and cell–cell adhesion; however it is not known whether they control formation of vessel lumens, which are essential for blood flow. Here, using an organotypic system that recapitulates distinct stages of VEGF-dependent angiogenesis, we show that lumen formation requires early cytoskeletal remodelling and lateral cell–cell contacts, mediated through the RAC1 guanine nucleotide exchange factor (GEF) DOCK4 (dedicator of cytokinesis 4). DOCK4 signalling is necessary for lateral filopodial protrusions and tubule remodelling prior to lumen formation, whereas proximal, tip filopodia persist in the absence of DOCK4. VEGF-dependent Rac activation via DOCK4 is necessary for CDC42 activation to signal filopodia formation and depends on the activation of RHOG through the RHOG GEF, SGEF. VEGF promotes interaction of DOCK4 with the CDC42 GEF DOCK9. These studies identify a novel Rho-family GTPase activation cascade for the formation of endothelial cell filopodial protrusions necessary for tubule remodelling, thereby influencing subsequent stages of lumen morphogenesis. 1 Institute of Cancer Research, Division of Cancer Biology, 237 Fulham Road, London SW3 6JB, UK. -

Cellular and Molecular Signatures in the Disease Tissue of Early
Cellular and Molecular Signatures in the Disease Tissue of Early Rheumatoid Arthritis Stratify Clinical Response to csDMARD-Therapy and Predict Radiographic Progression Frances Humby1,* Myles Lewis1,* Nandhini Ramamoorthi2, Jason Hackney3, Michael Barnes1, Michele Bombardieri1, Francesca Setiadi2, Stephen Kelly1, Fabiola Bene1, Maria di Cicco1, Sudeh Riahi1, Vidalba Rocher-Ros1, Nora Ng1, Ilias Lazorou1, Rebecca E. Hands1, Desiree van der Heijde4, Robert Landewé5, Annette van der Helm-van Mil4, Alberto Cauli6, Iain B. McInnes7, Christopher D. Buckley8, Ernest Choy9, Peter Taylor10, Michael J. Townsend2 & Costantino Pitzalis1 1Centre for Experimental Medicine and Rheumatology, William Harvey Research Institute, Barts and The London School of Medicine and Dentistry, Queen Mary University of London, Charterhouse Square, London EC1M 6BQ, UK. Departments of 2Biomarker Discovery OMNI, 3Bioinformatics and Computational Biology, Genentech Research and Early Development, South San Francisco, California 94080 USA 4Department of Rheumatology, Leiden University Medical Center, The Netherlands 5Department of Clinical Immunology & Rheumatology, Amsterdam Rheumatology & Immunology Center, Amsterdam, The Netherlands 6Rheumatology Unit, Department of Medical Sciences, Policlinico of the University of Cagliari, Cagliari, Italy 7Institute of Infection, Immunity and Inflammation, University of Glasgow, Glasgow G12 8TA, UK 8Rheumatology Research Group, Institute of Inflammation and Ageing (IIA), University of Birmingham, Birmingham B15 2WB, UK 9Institute of