Naja Vergani Triagem Funcional De Genes Envolvidos No
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-
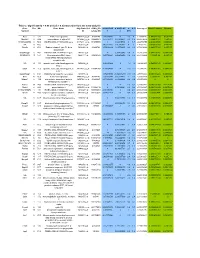
Table 2. Significant
Table 2. Significant (Q < 0.05 and |d | > 0.5) transcripts from the meta-analysis Gene Chr Mb Gene Name Affy ProbeSet cDNA_IDs d HAP/LAP d HAP/LAP d d IS Average d Ztest P values Q-value Symbol ID (study #5) 1 2 STS B2m 2 122 beta-2 microglobulin 1452428_a_at AI848245 1.75334941 4 3.2 4 3.2316485 1.07398E-09 5.69E-08 Man2b1 8 84.4 mannosidase 2, alpha B1 1416340_a_at H4049B01 3.75722111 3.87309653 2.1 1.6 2.84852656 5.32443E-07 1.58E-05 1110032A03Rik 9 50.9 RIKEN cDNA 1110032A03 gene 1417211_a_at H4035E05 4 1.66015788 4 1.7 2.82772795 2.94266E-05 0.000527 NA 9 48.5 --- 1456111_at 3.43701477 1.85785922 4 2 2.8237185 9.97969E-08 3.48E-06 Scn4b 9 45.3 Sodium channel, type IV, beta 1434008_at AI844796 3.79536664 1.63774235 3.3 2.3 2.75319499 1.48057E-08 6.21E-07 polypeptide Gadd45gip1 8 84.1 RIKEN cDNA 2310040G17 gene 1417619_at 4 3.38875643 1.4 2 2.69163229 8.84279E-06 0.0001904 BC056474 15 12.1 Mus musculus cDNA clone 1424117_at H3030A06 3.95752801 2.42838452 1.9 2.2 2.62132809 1.3344E-08 5.66E-07 MGC:67360 IMAGE:6823629, complete cds NA 4 153 guanine nucleotide binding protein, 1454696_at -3.46081884 -4 -1.3 -1.6 -2.6026947 8.58458E-05 0.0012617 beta 1 Gnb1 4 153 guanine nucleotide binding protein, 1417432_a_at H3094D02 -3.13334396 -4 -1.6 -1.7 -2.5946297 1.04542E-05 0.0002202 beta 1 Gadd45gip1 8 84.1 RAD23a homolog (S. -

The Inactive X Chromosome Is Epigenetically Unstable and Transcriptionally Labile in Breast Cancer
Supplemental Information The inactive X chromosome is epigenetically unstable and transcriptionally labile in breast cancer Ronan Chaligné1,2,3,8, Tatiana Popova1,4, Marco-Antonio Mendoza-Parra5, Mohamed-Ashick M. Saleem5 , David Gentien1,6, Kristen Ban1,2,3,8, Tristan Piolot1,7, Olivier Leroy1,7, Odette Mariani6, Hinrich Gronemeyer*5, Anne Vincent-Salomon*1,4,6,8, Marc-Henri Stern*1,4,6 and Edith Heard*1,2,3,8 Extended Experimental Procedures Cell Culture Human Mammary Epithelial Cells (HMEC, Invitrogen) were grown in serum-free medium (HuMEC, Invitrogen). WI- 38, ZR-75-1, SK-BR-3 and MDA-MB-436 cells were grown in Dulbecco’s modified Eagle’s medium (DMEM; Invitrogen) containing 10% fetal bovine serum (FBS). DNA Methylation analysis. We bisulfite-treated 2 µg of genomic DNA using Epitect bisulfite kit (Qiagen). Bisulfite converted DNA was amplified with bisulfite primers listed in Table S3. All primers incorporated a T7 promoter tag, and PCR conditions are available upon request. We analyzed PCR products by MALDI-TOF mass spectrometry after in vitro transcription and specific cleavage (EpiTYPER by Sequenom®). For each amplicon, we analyzed two independent DNA samples and several CG sites in the CpG Island. Design of primers and selection of best promoter region to assess (approx. 500 bp) were done by a combination of UCSC Genome Browser (http://genome.ucsc.edu) and MethPrimer (http://www.urogene.org). All the primers used are listed (Table S3). NB: MAGEC2 CpG analysis have been done with a combination of two CpG island identified in the gene core. Analysis of RNA allelic expression profiles (based on Human SNP Array 6.0) DNA and RNA hybridizations were normalized by Genotyping console. -
![Downloaded from [266]](https://docslib.b-cdn.net/cover/7352/downloaded-from-266-347352.webp)
Downloaded from [266]
Patterns of DNA methylation on the human X chromosome and use in analyzing X-chromosome inactivation by Allison Marie Cotton B.Sc., The University of Guelph, 2005 A THESIS SUBMITTED IN PARTIAL FULFILLMENT OF THE REQUIREMENTS FOR THE DEGREE OF DOCTOR OF PHILOSOPHY in The Faculty of Graduate Studies (Medical Genetics) THE UNIVERSITY OF BRITISH COLUMBIA (Vancouver) January 2012 © Allison Marie Cotton, 2012 Abstract The process of X-chromosome inactivation achieves dosage compensation between mammalian males and females. In females one X chromosome is transcriptionally silenced through a variety of epigenetic modifications including DNA methylation. Most X-linked genes are subject to X-chromosome inactivation and only expressed from the active X chromosome. On the inactive X chromosome, the CpG island promoters of genes subject to X-chromosome inactivation are methylated in their promoter regions, while genes which escape from X- chromosome inactivation have unmethylated CpG island promoters on both the active and inactive X chromosomes. The first objective of this thesis was to determine if the DNA methylation of CpG island promoters could be used to accurately predict X chromosome inactivation status. The second objective was to use DNA methylation to predict X-chromosome inactivation status in a variety of tissues. A comparison of blood, muscle, kidney and neural tissues revealed tissue-specific X-chromosome inactivation, in which 12% of genes escaped from X-chromosome inactivation in some, but not all, tissues. X-linked DNA methylation analysis of placental tissues predicted four times higher escape from X-chromosome inactivation than in any other tissue. Despite the hypomethylation of repetitive elements on both the X chromosome and the autosomes, no changes were detected in the frequency or intensity of placental Cot-1 holes. -

1 Supporting Information for a Microrna Network Regulates
Supporting Information for A microRNA Network Regulates Expression and Biosynthesis of CFTR and CFTR-ΔF508 Shyam Ramachandrana,b, Philip H. Karpc, Peng Jiangc, Lynda S. Ostedgaardc, Amy E. Walza, John T. Fishere, Shaf Keshavjeeh, Kim A. Lennoxi, Ashley M. Jacobii, Scott D. Rosei, Mark A. Behlkei, Michael J. Welshb,c,d,g, Yi Xingb,c,f, Paul B. McCray Jr.a,b,c Author Affiliations: Department of Pediatricsa, Interdisciplinary Program in Geneticsb, Departments of Internal Medicinec, Molecular Physiology and Biophysicsd, Anatomy and Cell Biologye, Biomedical Engineeringf, Howard Hughes Medical Instituteg, Carver College of Medicine, University of Iowa, Iowa City, IA-52242 Division of Thoracic Surgeryh, Toronto General Hospital, University Health Network, University of Toronto, Toronto, Canada-M5G 2C4 Integrated DNA Technologiesi, Coralville, IA-52241 To whom correspondence should be addressed: Email: [email protected] (M.J.W.); yi- [email protected] (Y.X.); Email: [email protected] (P.B.M.) This PDF file includes: Materials and Methods References Fig. S1. miR-138 regulates SIN3A in a dose-dependent and site-specific manner. Fig. S2. miR-138 regulates endogenous SIN3A protein expression. Fig. S3. miR-138 regulates endogenous CFTR protein expression in Calu-3 cells. Fig. S4. miR-138 regulates endogenous CFTR protein expression in primary human airway epithelia. Fig. S5. miR-138 regulates CFTR expression in HeLa cells. Fig. S6. miR-138 regulates CFTR expression in HEK293T cells. Fig. S7. HeLa cells exhibit CFTR channel activity. Fig. S8. miR-138 improves CFTR processing. Fig. S9. miR-138 improves CFTR-ΔF508 processing. Fig. S10. SIN3A inhibition yields partial rescue of Cl- transport in CF epithelia. -

Transcription Precedes Loss of Xist Coating and Depletion of H3k27me3 During X-Chromosome Reprogramming in the Mouse Inner Cell Mass Lucy H
RESEARCH ARTICLE 2049 Development 138, 2049-2057 (2011) doi:10.1242/dev.061176 © 2011. Published by The Company of Biologists Ltd Transcription precedes loss of Xist coating and depletion of H3K27me3 during X-chromosome reprogramming in the mouse inner cell mass Lucy H. Williams, Sundeep Kalantry*, Joshua Starmer and Terry Magnuson† SUMMARY Repression of Xist RNA expression is considered a prerequisite to reversal of X-chromosome inactivation (XCI) in the mouse inner cell mass (ICM), and reactivation of X-linked genes is thought to follow loss of Xist RNA coating and heterochromatic markers of inactivation, such as methylation of histone H3. We analyzed X-chromosome activity in developing ICMs and show that reactivation of gene expression from the inactive-X initiates in the presence of Xist coating and H3K27me3. Furthermore, depletion of Xist RNA coating through forced upregulation of NANOG does not result in altered reactivation kinetics. Taken together, our observations suggest that in the ICM, X-linked gene transcription and Xist coating are uncoupled. These data fundamentally alter our perception of the reactivation process and support the existence of a mechanism to reactivate Xp-linked genes in the ICM that operates independently of loss of Xist RNA and H3K27me3 from the imprinted inactive-X. KEY WORDS: X inactivation, Epigenetic reprogramming, Pre-implantation mouse development INTRODUCTION gradually coats the Xp during pre-implantation development X-chromosome inactivation (XCI) results in the transcriptional (Huynh and Lee, 2003; Okamoto et al., 2004). By the early silencing of most genes on one of the two X-chromosomes to blastocyst stage, prior to specification of extra-embryonic and equalize X-linked gene dosage in female cells with that in XY male embryonic lineages, Xist accumulation on the Xp (Xp-Xist coating) cells (Avner and Heard, 2001). -

Karla Alejandra Vizcarra Zevallos Análise Da Função De Genes
Karla Alejandra Vizcarra Zevallos Análise da função de genes candidatos à manutenção da inativação do cromossomo X em humanos Dissertação apresentada ao Pro- grama de Pós‐Graduação Inter- unidades em Biotecnologia USP/ Instituto Butantan/ IPT, para obtenção do Título de Mestre em Ciências. São Paulo 2017 Karla Alejandra Vizcarra Zevallos Análise da função de genes candidatos à manutenção da inativação do cromossomo X em humanos Dissertação apresentada ao Pro- grama de Pós‐Graduação Inter- unidades em Biotecnologia do Instituto de Ciências Biomédicas USP/ Instituto Butantan/ IPT, para obtenção do Título de Mestre em Ciências. Área de concentração: Biotecnologia Orientadora: Profa. Dra. Lygia da Veiga Pereira Carramaschi Versão corrigida. A versão original eletrônica encontra-se disponível tanto na Biblioteca do ICB quanto na Biblioteca Digital de Teses e Dissertações da USP (BDTD) São Paulo 2017 UNIVERSIDADE DE SÃO PAULO Programa de Pós-Graduação Interunidades em Biotecnologia Universidade de São Paulo, Instituto Butantan, Instituto de Pesquisas Tecnológicas Candidato(a): Karla Alejandra Vizcarra Zevallos Título da Dissertação: Análise da função de genes candidatos à manutenção da inativação do cromossomo X em humanos Orientador: Profa. Dra. Lygia da Veiga Pereira Carramaschi A Comissão Julgadora dos trabalhos de Defesa da Dissertação de Mestrado, em sessão pública realizada a ........./......../.........., considerou o(a) candidato(a): ( ) Aprovado(a) ( ) Reprovado(a) Examinador(a): Assinatura: .............................................................................. -

Paternal RLIM/Rnf12 Is a Survival Factor for Milk-Producing Alveolar Cells
Paternal RLIM/Rnf12 Is a Survival Factor for Milk-Producing Alveolar Cells Baowei Jiao,1 Hong Ma,1 Maxim N. Shokhirev,5 Alexander Drung,1 Qin Yang,1 JongDae Shin,1 Shaolei Lu,2 Meg Byron,3 Sundeep Kalantry,6 Arthur M. Mercurio,2 Jeanne B. Lawrence,3 Alexander Hoffmann,5 and Ingolf Bach1,4,* 1Program in Gene Function and Expression 2Department of Cancer Biology 3Department of Cell Biology 4Program in Molecular Medicine University of Massachusetts Medical School, Worcester, MA 01605, USA 5BioCircuits Institute, San Diego Center for Systems Biology of Cellular Stress Responses and Program in Bioinformatics and Systems Biology, University of California, San Diego, La Jolla, CA 92093, USA 6Department of Human Genetics, University of Michigan Medical School, Ann Arbor, MI 48109, USA *Correspondence: [email protected] DOI 10.1016/j.cell.2012.02.056 SUMMARY a bias to silence the paternal X chromosome (Gregg et al., 2010; Wang et al., 2010), although the physiological importance In female mouse embryos, somatic cells undergo of this bias is unclear. a random form of X chromosome inactivation (XCI), Milk-producing alveolar cells in the mammary gland are whereas extraembryonic trophoblast cells in the generated during alveolar morphogenesis in pregnant female placenta undergo imprinted XCI, silencing exclu- mice (Visvader, 2009). Prolactin (Prl) signaling via Jak2 and sively the paternal X chromosome. Initiation of im- Stat5 is essential for the differentiation and expansion of alveolar printed XCI requires a functional maternal allele of cells in pregnant and lactating females (Hennighausen and Rob- inson, 2008). Upon weaning, mammary alveolar cells undergo the X-linked gene Rnf12, which encodes the ubiquitin apoptosis in a process called involution (Sutherland et al., ligase Rnf12/RLIM. -

Pathogenic Variants in E3 Ubiquitin Ligase RLIM/RNF12 Lead to a Syndromic X-Linked Intellectual Disability and Behavior Disorder
Molecular Psychiatry https://doi.org/10.1038/s41380-018-0065-x ARTICLE Pathogenic variants in E3 ubiquitin ligase RLIM/RNF12 lead to a syndromic X-linked intellectual disability and behavior disorder 1,2 3 4 5 6,7 Suzanna G. M. Frints ● Aysegul Ozanturk ● Germán Rodríguez Criado ● Ute Grasshoff ● Bas de Hoon ● 8 9,10 11,12 13 14 Michael Field ● Sylvie Manouvrier-Hanu ● Scott E. Hickey ● Molka Kammoun ● Karen W. Gripp ● 5 5 15,16 11,12,17 Claudia Bauer ● Christopher Schroeder ● Annick Toutain ● Theresa Mihalic Mosher ● 17 12,17 5 6 3 Benjamin J. Kelly ● Peter White ● Andreas Dufke ● Eveline Rentmeester ● Sungjin Moon ● 12,17 1,2 18 19 18 Daniel C Koboldt ● Kees E. P. van Roozendaal ● Hao Hu ● Stefan A. Haas ● Hans-Hilger Ropers ● 8 20,21 20 20 22 21 Lucinda Murray ● Eric Haan ● Marie Shaw ● Renee Carroll ● Kathryn Friend ● Jan Liebelt ● 22 23 1,2 13 13 Lynne Hobson ● Marjan De Rademaeker ● Joep Geraedts ● Jean-Pierre Fryns ● Joris Vermeesch ● 15,16 5 6 3 13 5 Martine Raynaud ● Olaf Riess ● Joost Gribnau ● Nicholas Katsanis ● Koen Devriendt ● Peter Bauer ● 20,24 3,25 6 26 Jozef Gecz ● Christelle Golzio ● Cristina Gontan ● Vera M. Kalscheuer Received: 23 November 2017 / Accepted: 28 February 2018 © Macmillan Publishers Limited, part of Springer Nature 2018 Abstract 1234567890();,: 1234567890();,: RLIM, also known as RNF12, is an X-linked E3 ubiquitin ligase acting as a negative regulator of LIM-domain containing transcription factors and participates in X-chromosome inactivation (XCI) in mice. We report the genetic and clinical findings of 84 individuals from nine unrelated families, eight of whom who have pathogenic variants in RLIM (RING finger LIM domain-interacting protein). -

Failure of Extra-Embryonic Progenitor Maintenance in the Absence of Dosage Compensation Joshua W
2130 RESEARCH ARTICLE DEVELOPMENT AND STEM CELLS Development 139, 2130-2138 (2012) doi:10.1242/dev.076497 © 2012. Published by The Company of Biologists Ltd Failure of extra-embryonic progenitor maintenance in the absence of dosage compensation Joshua W. Mugford, Della Yee and Terry Magnuson* SUMMARY Proper regulation of X-linked gene expression, termed dosage compensation, is required for the normal development of mammalian embryos. Through the process of X chromosome inactivation (XCI), somatic cells of mammalian females inactivate one of their two X chromosomes in order to balance X-linked gene dosage with their male counterparts. The process of XCI is dependent upon the long non-coding RNA Xist, which is expressed from and coats the inactivated X chromosome (Xi) in cis. During mouse embryogenesis, imprinted XCI inactivates the paternally inherited X chromosome (Xp) within the extra-embryonic lineages. Consequently, females harboring a paternally derived Xist mutation (X/XXist–) die owing to failure of imprinted XCI and, presumably, poor trophoblast development. Here, we investigate the consequence of two active X chromosomes in the extra- embryonic ectoderm (ExE) of X/XXist– female embryos. At embryonic day (E) 6.5, we find that the X/XXist– ExE lacks the transcriptional regulator CDX2, a factor required to maintain the ExE in a progenitor state. In addition, spongiotrophoblast progenitors are not maintained. Surprisingly, we observe evidence of an Xi in a subpopulation of X/XXist– ExE cells. We demonstrate further that trophectodermal stem cells derived from X/XXist– embryos completely reverse normal imprinted XCI patterns. Taken together, our data suggest that, much like in the cells of the epiblast, the initial imprint that establishes imprinted XCI is probably erased in ExE cells. -

Chromatin Condensation of Xist Genomic Loci During Oogenesis In
© 2015. Published by The Company of Biologists Ltd | Development (2015) 142, 4049-4055 doi:10.1242/dev.127308 RESEARCH REPORT Chromatin condensation of Xist genomic loci during oogenesis in mice Atsushi Fukuda1, Atsushi Mitani1,2, Toshiyuki Miyashita2, Akihiro Umezawa1 and Hidenori Akutsu1,3,* ABSTRACT (Augui et al., 2011; Sado and Sakaguchi, 2013; Nesterova et al., Repression of maternal Xist (Xm-Xist) during preimplantation in 2001). This expression pattern leads to the establishment of mouse embryos is essential for establishing imprinted X chromosome imprinted XCI in extra-embryonic tissues (Takagi and Sasaki, Xist Xist inactivation. Nuclear transplantation (NT) studies using nuclei derived 1975). Paternal (Xp- ) expression is driven by the deposition – from non-growing (ng) and full-grown (fg) oocytes have indicated that of maternal Rnf12 (also known as Rlim Mouse Genome maternal-specific repressive modifications are imposed on Xm-Xist Informatics) (Shin et al., 2010; Jonkers et al., 2009). However, Xist during oogenesis, as well as on autosomal imprinted genes. Recent the locus on the maternal X chromosome (Xm) is tightly studies have revealed that histone H3 lysine 9 trimethylation protected by epigenetic factors. Using parthenogenetic embryos, (H3K9me3) enrichments on Xm-Xist promoter regions are involved which are composed of two maternal genomes, we previously in silencing at the preimplantation stages. However, whether demonstrated that histone 3 lysine 9 trimethylation (H3K9me3) is Xist H3K9me3 is imposed on Xm-Xist during oogenesis is not known. essential for Xm- repression during early preimplantation Here, we dissected the chromatin states in ng and fg oocytes and phases (Fukuda et al., 2014). early preimplantation stage embryos. -

A Novel RLIM/RNF12 Variant Disrupts Protein Stability and Function To
www.nature.com/scientificreports OPEN A novel RLIM/RNF12 variant disrupts protein stability and function to cause severe Tonne–Kalscheuer syndrome Francisco Bustos1,9, Carmen Espejo‑Serrano1,9, Anna Segarra‑Fas1,9, Rachel Toth1, Alison J. Eaton2, Kristin D. Kernohan3,4, Meredith J. Wilson5,6, Lisa G. Riley7,8* & Greg M. Findlay1* Tonne–Kalscheuer syndrome (TOKAS) is an X‑linked intellectual disability syndrome associated with variable clinical features including craniofacial abnormalities, hypogenitalism and diaphragmatic hernia. TOKAS is caused exclusively by variants in the gene encoding the E3 ubiquitin ligase gene RLIM, also known as RNF12. Here we report identifcation of a novel RLIM missense variant, c.1262A>G p.(Tyr421Cys) adjacent to the regulatory basic region, which causes a severe form of TOKAS resulting in perinatal lethality by diaphragmatic hernia. Inheritance and X‑chromosome inactivation patterns implicate RLIM p.(Tyr421Cys) as the likely pathogenic variant in the afected individual and within the kindred. We show that the RLIM p.(Tyr421Cys) variant disrupts both expression and function of the protein in an embryonic stem cell model. RLIM p.(Tyr421Cys) is correctly localised to the nucleus, but is readily degraded by the proteasome. The RLIM p.(Tyr421Cys) variant also displays signifcantly impaired E3 ubiquitin ligase activity, which interferes with RLIM function in Xist long‑non‑coding RNA induction that initiates imprinted X‑chromosome inactivation. Our data uncover a highly disruptive missense variant in RLIM that causes a severe form of TOKAS, thereby expanding our understanding of the molecular and phenotypic spectrum of disease severity. Tonne–Kalscheuer syndrome (TOKAS; MIM #300978) is a recently described X-linked recessive multiple con- genital anomaly disorder1–3. -

E L If D a R B U K a Ist a N B U L Ü N Ive R Sit E Si Sa Ğ . B Il. E N St
ELIF DARBUKA İSTANBUL ÜNİVERSİTESİ SAĞ. BİL. ENST. YÜKSEK LİSANS TEZİ İSTANBUL-2018 T.C. İSTANBUL ÜNİVERSİTESİ SAĞLIK BİLİMLERİ ENSTİTÜSÜ ( YÜKSEK LİSANS TEZİ ) KOLOREKTAL KANSERLİ HASTALARDA TCEAL7 GENİNİN ARAŞTIRILMASI ELİF DARBUKA DANIŞMAN PROF. DR. ALİ NUR TURGUT ULUTİN GENETİK ANABİLİM DALI GENETİK PROGRAMI İSTANBUL-2018 ii TEZ ONAYI iii BEYAN Bu tez çalışmasının kendi çalışmam olduğunu, tezin planlanmasından yazımına kadar bütün safhalarda etik dışı davranışımın olmadığını, bu tezdeki bütün bilgileri akademik ve etik kurallar içinde elde ettiğimi, bu tez çalışmasıyla elde edilmeyen bütün bilgi ve yorumlara kaynak gösterdiğimi ve bu kaynakları da kaynaklar listesine aldığımı, yine bu tezin çalışılması ve yazımı sırasında patent ve telif haklarını ihlal edici bir davranışımın olmadığı beyan ederim. Elif DARBUKA iv İTHAF Tez çalışmamı; Aileme ithaf ediyorum… v TEŞEKKÜR Tez süresince ilgisini ve desteğini esirgemeyen tez danışmanım Prof. Dr.Turgut ULUTİN’e, Tez çalışmam ve yüksek lisans eğitimim boyunca değerli bilgilerini, katkılarını ve sabrını esirgemeyen, her konuda tecrübesine sonuna kadar güvendiğim değerli hocam Prof. Dr. Nur BUYRU’ya, Tez çalışmamın gerçekleşmesi için gerekli dokuların temin edilmesini sağlayan ve bu süreçte bana destek olan Uzm. Dr. Süleyman DEMİRYAS’a ve patolojik incelemelerde uzmanlığından faydalandığım Uzm. Dr. Nuray KEPİL’e Moleküler Genetik Laboratuvarı’na ilk adımımı attığımdan beri her konuda yardımcı olan, yol gösteren ve sabırla destek veren Doç Dr. Onur BAYKARA’ya, Dr. Seda Ekizoğlu ERATAK’a, Dr. Filiz ÖZDEMİR’e, MSc. Asuman ÇELEBİ’ye ve Dr. Didem SEVEN’e, Doç Dr. Hikmet KÖSEOĞLU’na, Laboratuvar ve yüksek lisans eğitim arkadaşlığından öte benim için dost olan, MSc. Aslı KARACAN’a, BSc. Pelin BULUT’a, BSc. Ceren ORHAN’a, MSc.