(Arecaceae): Citogenética, Contenido De Adn Y Análisis Moleculares Poblacionales
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

Análisis Aeropalinológico Del Parque Nacional El Palmar
Bol. Soc. Argent. Bot. 52 (3) 2017 N. E. Muñoz et al. - Análisis aeropalinológico del Parque NacionalISSN El0373-580 Palmar X Bol. Soc. Argent. Bot. 52 (3): 473-496. 2017 ANÁLISIS AEROPALINOLÓGICO EN TRES ÁREAS DE VEGETACIÓN DENTRO DEL PARQUE NACIONAL EL PALMAR (COLÓN, ENTRE RÍOS) Y SU RELACIÓN CON LA VEGETACIÓN LOCAL Y REGIONAL NADIA E. MUÑOZ1, MERCEDES DI PASQUO1, FERNANDO BIGANZOLI2 y WILLIAM B. BATISTA2,3 Summary: Aeropalinological analysis in three vegetation areas within El Palmar National Park (Colón, Entre Ríos) and its relationship with the local and regional vegetation. The diversity of pollen rain monthly collected during two years (2011-2013) from the atmosphere in Tauber traps located at three sites in El Palmar National Park (Entre Ríos Province) is used to characterize the source vegetation. Site 1 is a mixed area composed of grassland, palm savanna, and wetland communities, site 2 is a grassland area and site 3 is a dense palm savanna. A total of 71 pollen-grain types grouped in 43 families coming from local, regional and extra- regional areas are identified. Of them, sixteen pollen types with more than 1% of Annual Pollen Influx in at least two samples were used in this analysis. Different factors involved in quali-quantitave changes of taxa during the observation interval (e.g. pollination affinity, origin of pollen grains, canopy effect, meteorological variables) are further considered. The floral composition of each site compared to their palynoassemblages revealed that site 2 is characterized by a high abundance of Asteraeceae-Asteroideae, with an increase in the value of Vernonia (Asteraceae Cichoroidea) and Lamiaceae during the second year. -
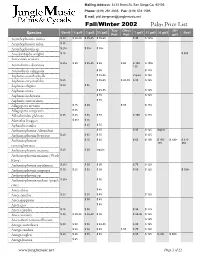
Winter-Fall Sale 2002 Palm Trees-Web
Mailing Address: 3233 Brant St. San Diego Ca, 92103 Phone: (619) 291 4605 Fax: (619) 574 1595 E mail: [email protected] Fall/Winter 2002 Palm Price List Tree Citrus 25/+ Band$ 1 gal$ 2 gal$ 3/5 gal$ 7 gal$ 15 gal$ 20 gal$ Box$ Species Pot$ Pot$ gal$ Acanthophoenix crinita $ 30 $ 30-40 $ 35-45 $ 55-65 $ 95 $ 125+ Acanthophoenix rubra $ 35 Acanthophoenix sp. $ 25+ $ 35+ $ 55+ Acoelorrhaphe wrightii $ 15 $ 300 Acrocomia aculeata $ 25+ $ 35 $ 35-45 $ 65 $ 65 $ 100- $ 150+ Actinokentia divaricata 135 Actinorhytis calapparia $ 55 $ 125 Aiphanes acanthophylla $ 45-55 inquire $ 125 Aiphanes caryotaefolia $ 25 $ 55-65 $ 45-55 $ 85 $ 125 Aiphanes elegans $ 20 $ 35 Aiphanes erosa $ 45-55 $ 125 Aiphanes lindeniana $ 55 $ 125 Aiphanes vincentsiana $ 55 Allagoptera arenaria $ 25 $ 40 $ 55 $ 135 Allagoptera campestris $ 35 Alloschmidtia glabrata $ 35 $ 45 $ 55 $ 85 $ 150 $ 175 Alsmithia longipes $ 35+ $ 55 Aphandra natalia $ 35 $ 55 Archontophoenix Alexandrae $ 55 $ 85 $ 125 inquire Archontophoenix Beatricae $ 20 $ 35 $ 55 $ 125 Archontophoenix $ 25 $ 45 $ 65 $ 100 $ 150- $ 200+ $ 310- 175 350 cunninghamiana Archontophoenix maxima $ 25 $ 30 inquire Archontophoenix maxima (Wash River) Archontophoenix myolaensis $ 25+ $ 30 $ 50 $ 75 $ 125 Archontophoenix purpurea $ 30 $ 25 $ 35 $ 50 $ 85 $ 125 $ 300+ Archontophoenix sp. Archontophoenix tuckerii (peach $ 25+ $ 55 river) Areca alicae $ 45 Areca catechu $ 20 $ 35 $ 45 $ 125 Areca guppyana $ 30 $ 45 Areca ipot $ 45 Areca triandra $ 25 $ 30 $ 95 $ 125 Areca vestiaria $ 25 $ 30-35 $ 35-40 $ 55 $ 85-95 $ 125 Arecastrum romanzoffianum $ 125 Arenga australasica $ 20 $ 30 $ 35 $ 45-55 $ 85 $ 125 Arenga caudata $ 20 $ 30 $ 45 $ 55 $ 75 $ 100 Arenga engleri $ 20 $ 60 $ 35 $ 45 $ 85 $ 125 $ 200 $ 300+ Arenga hastata $ 25 www.junglemusic.net Page 1 of 22 Tree Citrus 25/+ Band$ 1 gal$ 2 gal$ 3/5 gal$ 7 gal$ 15 gal$ 20 gal$ Box$ Species Pot$ Pot$ gal$ Arenga hookeriana inquire Arenga micranthe 'Lhutan' $ 20 inquire Arenga pinnata $ 35 $ 50 $ 85 $ 125 Arenga sp. -

Redalyc.ECOLOGICAL STRATEGIES and IMPACT of WILD BOAR IN
Mastozoología Neotropical ISSN: 0327-9383 [email protected] Sociedad Argentina para el Estudio de los Mamíferos Argentina Cuevas, M. Fernanda; Ojeda, Ricardo A.; Jaksic, Fabian M. ECOLOGICAL STRATEGIES AND IMPACT OF WILD BOAR IN PHYTOGEOGRAPHIC PROVINCES OF ARGENTINA WITH EMPHASIS ON ARIDLANDS Mastozoología Neotropical, vol. 23, núm. 2, 2016, pp. 239-254 Sociedad Argentina para el Estudio de los Mamíferos Tucumán, Argentina Available in: http://www.redalyc.org/articulo.oa?id=45750282004 How to cite Complete issue Scientific Information System More information about this article Network of Scientific Journals from Latin America, the Caribbean, Spain and Portugal Journal's homepage in redalyc.org Non-profit academic project, developed under the open access initiative Mastozoología Neotropical, 23:239-254, Mendoza, 2016 Copyright ©SAREM, 2016 http://www.sarem.org.ar Versión impresa ISSN 0327-9383 http://www.sbmz.com.br Versión on-line ISSN 1666-0536 Sección Especial MAMÍFEROS EXÓTICOS INVASORES ECOLOGICAL STRATEGIES AND IMPACT OF WILD BOAR IN PHYTOGEOGRAPHIC PROVINCES OF ARGENTINA WITH EMPHASIS ON ARIDLANDS M. Fernanda Cuevas1, Ricardo A. Ojeda1 and Fabian M. Jaksic2 1 Grupo de Investigaciones de la Biodiversidad (GiB), IADIZA, CCT Mendoza CONICET, CC 507, 5500 Mendoza, Argentina. [Correspondence: <[email protected]>]. 2 Centro de Ecología Aplicada y Sustentabilidad (CAPES), Pontificia Universidad Católica de Chile, Casilla 114-D, Santiago, Chile ABSTRACT. Wild boar is an invasive species introduced to Argentina for sport hunting purposes. Here, this spe- cies is present in at least 8 phytogeographic provinces but we only have information in four of them (Pampean grassland, Espinal, Subantarctic and Monte Desert). We review the ecological strategies and impact of wild boar on ecosystem processes in these different phytogeographic provinces and identify knowledge gaps and research priorities for a better understanding of this invasive species in Argentina. -

Sfps Fall 2011 Sale Plant List
SFPS FALL 2011 SALE PLANT LIST PLANTS VENDOR # Palms Acanthophoenix rubra 35 Acoelorrhaphe wrightii 26, 67 Acrocomia aculeata 50, 67 Actinokentia divaricata 35, 57, 66, 68, 72 Actinorhytis calapparia 72 Adonidia merrillii 31, 57, 66, 89 Adonidia merrillii var. "Golden Form" 35 Aiphanes aculeata = Aiphanes horrida - Aiphanes caryotifolia = Aiphanes horrida - Aiphanes erosa = Aiphanes minima - Aiphanes horrida 35, 68, 72 Aiphanes minima 68 Aiphanes vincentiana = Aiphanes minima - Allagoptera arenaria 57, 66, 67, 68, 72 Allagoptera campestris 67 Allagoptera leucocalyx 57 Alloschmidia glabrata = Basselinia glabrata - Alsmithia longipes = Heterospathe longipes - Archontophoenix cunninghamiana var. 'Illawara' 68 Archontophoenix maxima 67, 72 Archontophoenix myolensis 50, 66, 67, 68 Archontophoenix purpurea 57, 66, 72 Archontophoenix tuckeri 66, 68 Areca aliceae = Areca triandra - Areca camarinensis 57, 68 Areca catechu 57, 67, 72 Areca catechu var. 'Dwarf' 35, 50 Areca hutchinsoniana 68 Areca ipot 67 Areca latiloba = Areca montana - Areca macrocalyx var. 'Red Form' 35, 57, 68 Areca macrocarpa 68 Areca montana 57 Areca triandra 68, 72 Areca vestiaria 25, 35, 57, 67, 68 Areca vestiaria var. 'Orange Form' 25, 57, 67, 72 Areca vestiaria var. 'Maroon Leaf' 35, 57, 67 Areca vestiaria var. 'Red Leaf' 57, 67, 72 Areca sp. 'Yellow Crownshaft' 25 Arenga ambong = Arenga undulatifolia - Arenga brevipes 57 Arenga caudata 66 Arenga engleri 31, 66, 68, 72 Arenga hookeriana 35, 57, 66, 72 Arenga microcarpa 26, 66 Arenga obtusifolia 57, 66 PLANTS VENDOR # Arenga pinnata 50, 57, 66, 67, 68 Arenga porphyrocarpa 66 Arenga tremula 26, 57, 66, 68, 72 Arenga undulatifolia 35, 57, 66, 67 Arenga westerhoutii 68 Asterogyne martiana 57, 68, 72 Astrocaryum acaule 72 Astrocaryum alatum 35, 50, 57, 67 Astrocaryum mexicanum 72 Astrocaryum murumuru 72 Attalea butyracea 57, 67, 72 Attalea cohune 35 Attalea phalerata 50, 91 Attalea rostrata 68 Attalea speciosa 50, 66 Bactris bidentula 72 Bactris gasipaes 67 Bactris gasipaes var. -

Breeding Biology of the White-Winged Nightjar (Eleothreptus Candicans) in Eastern Paraguay
Revista Brasileira de Ornitologia, 22(2), 219-233 ARTICLE June 2014 Breeding biology of the White-winged Nightjar (Eleothreptus candicans) in eastern Paraguay Robert G. Pople Department of Zoology, University of Cambridge, Downing Street, Cambridge, CB2 3EJ, UK. Email: [email protected] Current address: BirdLife International, Wellbrook Court, Girton Road, Cambridge, CB3 0NA, UK. Received on 03 September 2013. Accepted on 02 October 2013. ABSTRACT: Breeding biology of the White-winged Nightjar (Eleothreptus candicans) in eastern Paraguay. I present the first detailed description of the breeding biology of the White-winged Nightjar (Eleothreptus candicans), based on data collected over three breeding seasons during 1998-2001 at Aguará Ñu, Canindeyú, eastern Paraguay. Male nightjars defended small territories situated on the upper slopes of ridgelines. Each territory contained one or more “display arenas” at which the male performed nuptial display flights. Aggregation indices confirmed that the primary display arenas of males were significantly clustered within the survey area. Within their territories, males apparently selected display arenas on the basis of their structural characteristics: mounds used as arenas were significantly lower and broader than random mounds. Males engaged in display activity from late August to early January. On average, males performed 0.54 ± 0.04 display flights per minute during nocturnal focal watches, but there was considerable intra-male variation in display rate. Following a burst of activity immediately after their arrival at display arenas at dusk, male display rate was best explained by ambient levels of moonlight. Males produced a previously undescribed insect-like “tik tik” call when inactive on their territories. -

Las Palmeras En El Marco De La Investigacion Para El
REVISTA PERUANA DE BIOLOGÍA Rev. peru: biol. ISSN 1561-0837 Volumen 15 Noviembre, 2008 Suplemento 1 Las palmeras en el marco de la investigación para el desarrollo en América del Sur Contenido Editorial 3 Las comunidades y sus revistas científicas 1he scienrific cornmuniries and their journals Leonardo Romero Presentación 5 Laspalmeras en el marco de la investigación para el desarrollo en América del Sur 1he palrns within the framework ofresearch for development in South America Francis Kahny CésarArana Trabajos originales 7 Laspalmeras de América del Sur: diversidad, distribución e historia evolutiva 1he palms ofSouth America: diversiry, disrriburíon and evolutionary history Jean-Christopbe Pintaud, Gloria Galeano, Henrik Balslev, Rodrigo Bemal, Fmn Borchseníus, Evandro Ferreira, Jean-Jacques de Gran~e, Kember Mejía, BettyMillán, Mónica Moraes, Larry Noblick, FredW; Staufl'er y Francis Kahn . 31 1he genus Astrocaryum (Arecaceae) El género Astrocaryum (Arecaceae) . Francis Kahn 49 1he genus Hexopetion Burret (Arecaceae) El género Hexopetion Burret (Arecaceae) Jean-Cbristopbe Pintand, Betty MiJJány Francls Kahn 55 An overview ofthe raxonomy ofAttalea (Arecaceae) Una visión general de la taxonomía de Attalea (Arecaceae) Jean-Christopbe Pintaud 65 Novelties in the genus Ceroxylon (Arecaceae) from Peru, with description ofa new species Novedades en el género Ceroxylon (Arecaceae) del Perú, con la descripción de una nueva especie Gloria Galeano, MariaJosé Sanín, Kember Mejía, Jean-Cbristopbe Pintaud and Betty MiJJán '73 Estatus taxonómico -

Projeto Mestrado
UNIVERSIDADE FEDERAL DO RIO GRANDE DO SUL FACULDADE DE AGRONOMIA PROGRAMA DE PÓS-GRADUAÇÃO EM FITOTECNIA CARACTERIZAÇÃO MORFOANATÔMICA E HISTOQUÍMICA DE SEMENTES DE Butia odorata (BARB. RODR.) NOBLICK DURANTE A SUPERAÇÃO DA DORMÊNCIA Bruna Baratto Bióloga/UNIVATES Dissertação apresentada como um dos requisitos à obtenção do Grau de Mestre em Fitotecnia Ênfase Fisiologia e Manejo Vegetal Porto Alegre (RS), Brasil Fevereiro de 2016 ii Folha de homologação iii À minha mãe, Maristela Baratto, ao meu pai, Odacir Baratto, e à minha amada avó, Rosina Baratto; Dedico. iv AGRADECIMENTOS Se estás lendo esta página, é porque eu consegui. E no final destes dois anos, construído por momentos felizes e outros nem tanto. Contei com o apoio de várias pessoas para alcançar esta vitória e por isso, deixo aqui meus mais sinceros agradecimentos... À DEUS, A CAPES pela concessão da bolsa. Ao meu orientador, Prof. Dr. Claudimar Sidnei Fior, pelo aceite da orientação, pela amizade, pelo incentivo, calma e bom humor nas horas mais dificeis da realização deste trabalho. Sem contar com os valiosos conhecimentos compartilhados e pelo excepcional talento em ensinar. À minha co-orientadora, Profª. Drª. Alexandra Antunes Mastroberti, pelos ensinamentos que foram essenciais para o desenvolvimento deste trabalho. Aos membros da banca examinadora, Adilson Tonietto, Sofia Aumond Kuhn e Gilmar Arduino Bettio Marodin, pela solicitude em participar da avaliação. Um agradecimento especial ao pesquisador Gilson Schlindwein, pela honra de sua parceria neste trabalho, além de compartilhar seus conhecimentos do início ao fim deste. Ao Prof. Dr. Jorge Ernesto Mariath, pela disponibilização de estrutura para a realização desta pesquisa, bem como me receber. -

From Barcelona to Bordighera: Palm Gardens on Mediterranean Shores
PALMS Pintaud: Mediterranean Palm Gardens Volume 46(3) 2002 From Barcelona JEAN-CHRISTOPHE PINTAUD to Bordighera: IRD, UMR DGPC Laboratoire GENETROP 911 Avenue Agropolis Palm Gardens on BP 64501 34394 Montpellier Mediterranean Cedex 5, France Shores 1. Native Chamaerops humilis south of Barcelona. The species occurs in large numbers on rocky limestone slopes in front of the sea. Palms and palm landscapes are acknowledged as symbols of exoticism and as such contribute greatly in attracting people, especially tourists, to the Mediterranean region. Thus there is a need to preserve the region’s many historical palm gardens. Municipalities play a very important role in the conservation of the palm heritage. Nice has a network of parks, mostly originating from ancient private properties, and is developing a new botanical garden. The cities of San Remo, Menton, Cannes, Hyères, Toulon and Le Pradet are developing comprehensive new palm collections in old, renovated gardens, often in association with the French Palm Society (Fous de Palmiers). PALMS 46(3): 149–153 149 PALMS Pintaud: Mediterranean Palm Gardens Volume 46(3) 2002 The northwestern part of the Mediterranean Sea cultivation, the date palm will completely and adjacent European continent have an disappear. unusually warm climate for this latitude Bordighera’s date palms also played an important (41–44°N). Palms reach the northern limit of their role in several respects in the 19th century natural distribution there, with the Mediterranean development of the Riviera. Early palm land- fan palm Chamaerops humilis. This species is a scaping was made with Bordighera’s palms, the typical component – an indicator – of the warmest only significant source of well-grown plants before Mediterranean vegetation zone. -

Diferenciação Polínica De Butia, Euterpe, Geonoma, Syagrus E Thritrinax E Implicações Paleoecológicas De Arecaceae Para O Rio Grande Do Sul
Diferenciação polínica de Butia, Euterpe, Genoma, Syagrus e Thritrinax ... 35 Diferenciação polínica de Butia, Euterpe, Geonoma, Syagrus e Thritrinax e implicações paleoecológicas de Arecaceae para o Rio Grande do Sul. Soraia Girardi Bauermann, Andréia Cardoso Pacheco Evaldt, Janaína Rosana Zanchin & Sergio Augusto de Loreto Bordignon Universidade Luterana do Brasil – Laboratório de Palinologia, Av. Farroupilha, 8001. Caixa Postal, 124, CEP. 92425-900, Canoas, RS, Brasil. [email protected] Recebido em 08.VI.2009. Aceito em 03.V.2010 RESUMO – As Arecaceae ou “palmeiras”, como são popularmente conhecidas, compreendem 207 gêneros e 2.675 espécies. Pouco é conhecido sobre sua história paleoecológica no extremo sul do Brasil, principalmente devido à difi culdade de separação das espécies no registro polínico. Para o Estado, é citada a ocorrência de 11 espécies, sendo que 9 são apresentadas neste trabalho contribuindo assim com dados inéditos desta família para o Rio Grande do Sul. A preparação dos grãos de pólen para posterior análise foi realizada através de acetólise. Fez-se descrição polínica dos grãos de pólen de Arecaceae baseado em seus atributos quanti e qualitativos. A análise morfológica das espécies mostrou grãos de pólen estenopolínicos, porém apresentando diferenças em relação ao tamanho e ornamentação, possibilitando o estabelecimento de quatro tipos polínicos. Através dos dados de distribuição e hábitat das espécies foi possível estabelecer correlação entre os tipos polínicos e o ambiente onde as plantas se desenvolvem. Palavras-chave: grãos de pólen, Palmae, morfologia polínica, Arecales. ABSTRACT – Pollen Difference in Butia, Euterpe, Geonoma, Syagrus and Thritrinax and paleoecological implications of Arecaceae for Rio Grande do Sul. The Arecaceae or “palm”, as they are popularly known, comprises 207 genera and 2675 species. -

Page 1 T H E M O N T G O M E R Y N E W S T H E M O N T G O M E R Y
THETHE MONTGOMERYMONTGOMERY NEWSNEWS Newsletter of Montgomery Botanical Center VOL. VOL. 12 NO. 1 SPRING SPRING 2004 WhoWho WeWe AreAre Terrence Walters, Ph.D. Executive Director Lee Anderson Manager, Horticulture & Facilities Mary Andrews Manager, Development & Communications A Botanical Garden Charles Bauduy Assistant Palm Horticulturist Jack Bauer Facilities Supervisor Barbara Bohnsack Built for Science Field Supervisor Mario Borroto Landscaper Terrence Walters, Ph.D. Juan Corona MBC Executive Director Equipment Specialist Orlando Coy Grounds Supervisor Stella Cuestas Assistant Cycad Horticulturist Laurie Danielson Palm Horticulturist Abbie Dasher Landscaper Willy Dye Landscaper Christine Emshousen Cycad Horticulturist Jody Haynes Cycad Biologist Barbara Judd Nursery Horticulturist Judith Kay Seedbank Coordinator Martha Lagos Housekeeper Scott Massey Dicot Horticulturist Vickie Murphy Assistant Palm Horticulturist Larry Noblick, Ph.D. Manager, Collections Development Palm Biologist Willie Payne Landscaper Jessie Pender Landscaper Annamaria Richcreek Administrator Randy Russ Landscaper Arantza Strader Database Assistant Ansel Thomas Irrigation Specialist Hostilio Torres This population of Caryota gigas in the MBC collection not only provides an attractive visual Equipment Operator Marino Valcourt presentation of shapes and textures, but, along with its associated data, is far more valuable Irrigation Technician to researchers than if just one or two representatives of the species were available. Laura Vasquez Field Specialist Evelyn -

Biosíntesi, Distribució, Acumulació I Funció De La Vitamina E En Llavors: Mecanismes De Control
Biosíntesi, distribució, acumulació i funció de la vitamina E en llavors: mecanismes de control Laura Siles Suárez Aquesta tesi doctoral està subjecta a la llicència Reconeixement- NoComercial – SenseObraDerivada 3.0. Espanya de Creative Commons. Esta tesis doctoral está sujeta a la licencia Reconocimiento - NoComercial – SinObraDerivada 3.0. España de Creative Commons. This doctoral thesis is licensed under the Creative Commons Attribution-NonCommercial- NoDerivs 3.0. Spain License. Barcelona, febrer de 2017 Biosíntesi, distribució, acumulació i funció de la vitamina E en llavors: mecanismes de control Memòria presentada per Laura Siles Suarez per a optar al grau de Doctora per la Universitat de Barcelona. Aquest treball s’emmarca dins el programa de doctorat de BIOLOGIA VEGETAL del Departament de Biologia Evolutiva, Ecologia i Ciències Ambientals (BEECA) de la Facultat de Biologia de la Universitat de Barcelona. El present treball ha estat realitzat al Departament de Biologia Evolutiva, Ecologia i Ciències Ambientals de la Facultat de Biologia (BEECA) de la Universitat de Barcelona sota la direcció de la Dra. Leonor Alegre Batlle i el Dr. Sergi Munné Bosch. Doctoranda: Directora i Codirector de Tesi: Tutora de Tesi: Laura Siles Suarez Dra. Leonor Alegre Batlle Dra. Leonor Alegre Batlle Dr. Sergi Munné Bosch “Mira profundamente en la naturaleza y entonces comprenderás todo mejor”- Albert Einstein. “La creación de mil bosques está en una bellota”-Ralph Waldo Emerson. A mi familia, por apoyarme siempre, y a mis bichejos peludos Índex ÍNDEX AGRAÏMENTS i ABREVIATURES v INTRODUCCIÓ GENERAL 1 Vitamina E 3 1.1.Descobriment i estudi 3 1.2.Estructura química i classes 3 Distribució de la vitamina E 5 Biosíntesi de vitamina E 6 3.1. -

Table of Contents Than a Proper TIMOTHY K
Palms Journal of the International Palm Society Vol. 57(1) Mar. 2013 PALMS Vol. 57(1) 2013 CONTENTS Island Hopping for Palms in Features 5 Micronesia D.R. H ODEL Palm News 4 Palm Literature 36 Shedding Light on the 24 Pseudophoenix Decline S. E DELMAN & J. R ICHARDS An Anatomical Character to 30 Support the Cohesive Unit of Butia Species C. M ARTEL , L. N OBLICK & F.W. S TAUFFER Phoenix dactylifera and P. sylvestris 37 in Northwestern India: A Glimpse of their Complex Relationships C. N EWTON , M. G ROS -B ALTHAZARD , S. I VORRA , L. PARADIS , J.-C. P INTAUD & J.-F. T ERRAL FRONT COVER A mighty Metroxylon amicarum , heavily laden with fruits and festooned with epiphytic ferns, mosses, algae and other plants, emerges from the low-hanging clouds near Nankurupwung in Nett, Pohnpei. See article by D.R. Hodel, p. 5. Photo by D.R. Hodel. The fruits of Pinanga insignis are arranged dichotomously BACK COVER and ripen from red to Hydriastele palauensis is a tall, slender palm with a whitish purplish black. See article by crownshaft supporting the distinctive canopy. See article by D.R. Hodel, p. 5. Photo by D.R. Hodel, p. 5. Photo by D.R. Hodel . D.R. Hodel. 3 PALMS Vol. 57(1) 2013 PALM NEWS Last year, the South American Palm Weevil ( Rhynchophorus palmarum ) was found during a survey of the Lower Rio Grande Valley, Texas . This palm-killing weevil has caused extensive damage in other parts of the world, according to Dr. Raul Villanueva, an entomologist at the Texas A&M AgriLife Research and Extension Center at Weslaco.