Maladies Rares Et" Big Data": Solutions Bioinformatiques Vers Une Analyse Guidée Par Les Connaissances: Applications Aux Ciliopathies
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

Constitutive Scaffolding of Multiple Wnt Enhanceosome Components By
RESEARCH ARTICLE Constitutive scaffolding of multiple Wnt enhanceosome components by Legless/ BCL9 Laurens M van Tienen, Juliusz Mieszczanek, Marc Fiedler, Trevor J Rutherford, Mariann Bienz* MRC Laboratory of Molecular Biology, Cambridge, United Kingdom Abstract Wnt/b-catenin signaling elicits context-dependent transcription switches that determine normal development and oncogenesis. These are mediated by the Wnt enhanceosome, a multiprotein complex binding to the Pygo chromatin reader and acting through TCF/LEF- responsive enhancers. Pygo renders this complex Wnt-responsive, by capturing b-catenin via the Legless/BCL9 adaptor. We used CRISPR/Cas9 genome engineering of Drosophila legless (lgs) and human BCL9 and B9L to show that the C-terminus downstream of their adaptor elements is crucial for Wnt responses. BioID proximity labeling revealed that BCL9 and B9L, like PYGO2, are constitutive components of the Wnt enhanceosome. Wnt-dependent docking of b-catenin to the enhanceosome apparently causes a rearrangement that apposes the BCL9/B9L C-terminus to TCF. This C-terminus binds to the Groucho/TLE co-repressor, and also to the Chip/LDB1-SSDP enhanceosome core complex via an evolutionary conserved element. An unexpected link between BCL9/B9L, PYGO2 and nuclear co-receptor complexes suggests that these b-catenin co-factors may coordinate Wnt and nuclear hormone responses. DOI: 10.7554/eLife.20882.001 *For correspondence: mb2@mrc- Introduction lmb.cam.ac.uk The Wnt/b-catenin signaling cascade is an ancient cell communication pathway that operates con- Competing interests: The text-dependent transcriptional switches to control animal development and tissue homeostasis authors declare that no (Cadigan and Nusse, 1997). -

Mouse Cep104 Conditional Knockout Project (CRISPR/Cas9)
https://www.alphaknockout.com Mouse Cep104 Conditional Knockout Project (CRISPR/Cas9) Objective: To create a Cep104 conditional knockout Mouse model (C57BL/6J) by CRISPR/Cas-mediated genome engineering. Strategy summary: The Cep104 gene (NCBI Reference Sequence: NM_177673 ; Ensembl: ENSMUSG00000039523 ) is located on Mouse chromosome 4. 22 exons are identified, with the ATG start codon in exon 2 and the TGA stop codon in exon 22 (Transcript: ENSMUST00000047497). Exon 5~6 will be selected as conditional knockout region (cKO region). Deletion of this region should result in the loss of function of the Mouse Cep104 gene. To engineer the targeting vector, homologous arms and cKO region will be generated by PCR using BAC clone RP23-101G23 as template. Cas9, gRNA and targeting vector will be co-injected into fertilized eggs for cKO Mouse production. The pups will be genotyped by PCR followed by sequencing analysis. Note: Exon 5 starts from about 15.41% of the coding region. The knockout of Exon 5~6 will result in frameshift of the gene. The size of intron 4 for 5'-loxP site insertion: 1291 bp, and the size of intron 6 for 3'-loxP site insertion: 1224 bp. The size of effective cKO region: ~1111 bp. The cKO region does not have any other known gene. Page 1 of 7 https://www.alphaknockout.com Overview of the Targeting Strategy Wildtype allele gRNA region 5' gRNA region 3' 1 3 4 5 6 7 8 22 Targeting vector Targeted allele Constitutive KO allele (After Cre recombination) Legends Exon of mouse Cep104 Homology arm cKO region loxP site Page 2 of 7 https://www.alphaknockout.com Overview of the Dot Plot Window size: 10 bp Forward Reverse Complement Sequence 12 Note: The sequence of homologous arms and cKO region is aligned with itself to determine if there are tandem repeats. -

Fish Mutant, Where Is Thy Phenotype?
PERSPECTIVE Fish mutant, where is thy phenotype? Darius Balciunas* Department of Biology, Temple University, Philadelphia, Pennsylvania, United States of America * [email protected] The field of genetics emerged as a study of the inheritance of desirable or otherwise interesting traits. Mutations were characterized as recessive or dominant, assigned to complementation groups, mapped, and finally, the affected genes were identified. By default, recessive mutations could only be isolated in genes that played an important role in the biological process of inter- est, be it pea color or segmentation pattern of the fruit fly embryo. As methodologies to mutate specific genes in various model systems were developed, scien- tists began to employ reverse genetics, whereby a gene of interest is selected and a mutant is generated. Ideally, the mutant displays a phenotype that can be studied (green panels in Fig 1). Such best-case scenarios pose the danger of confirmation bias ([1] and references therein). It may therefore be prudent to validate the phenotype by engineering an independent mutant allele. This is especially straightforward in zebrafish, given that targeted mutagenesis using CRISPR/Cas9 requires relatively little effort [2±4]. In zebrafish, the phenotype may also be confirmed by performing knockdown using morpholino oligonucleotides [5±6]. a1111111111 Real experiments rarely follow best-case scenarios, and very frequently, mutants generated a1111111111 by reverse genetics fail to display overt phenotypes. Are the majority of protein-coding genes a1111111111 indeed not required, often despite a very high degree of evolutionary conservation? Genetic a1111111111 redundancy, most obviously in the form of homologous or duplicated genes, certainly contrib- a1111111111 utes to a lack of mutant phenotypes. -

A Computational Approach for Defining a Signature of Β-Cell Golgi Stress in Diabetes Mellitus
Page 1 of 781 Diabetes A Computational Approach for Defining a Signature of β-Cell Golgi Stress in Diabetes Mellitus Robert N. Bone1,6,7, Olufunmilola Oyebamiji2, Sayali Talware2, Sharmila Selvaraj2, Preethi Krishnan3,6, Farooq Syed1,6,7, Huanmei Wu2, Carmella Evans-Molina 1,3,4,5,6,7,8* Departments of 1Pediatrics, 3Medicine, 4Anatomy, Cell Biology & Physiology, 5Biochemistry & Molecular Biology, the 6Center for Diabetes & Metabolic Diseases, and the 7Herman B. Wells Center for Pediatric Research, Indiana University School of Medicine, Indianapolis, IN 46202; 2Department of BioHealth Informatics, Indiana University-Purdue University Indianapolis, Indianapolis, IN, 46202; 8Roudebush VA Medical Center, Indianapolis, IN 46202. *Corresponding Author(s): Carmella Evans-Molina, MD, PhD ([email protected]) Indiana University School of Medicine, 635 Barnhill Drive, MS 2031A, Indianapolis, IN 46202, Telephone: (317) 274-4145, Fax (317) 274-4107 Running Title: Golgi Stress Response in Diabetes Word Count: 4358 Number of Figures: 6 Keywords: Golgi apparatus stress, Islets, β cell, Type 1 diabetes, Type 2 diabetes 1 Diabetes Publish Ahead of Print, published online August 20, 2020 Diabetes Page 2 of 781 ABSTRACT The Golgi apparatus (GA) is an important site of insulin processing and granule maturation, but whether GA organelle dysfunction and GA stress are present in the diabetic β-cell has not been tested. We utilized an informatics-based approach to develop a transcriptional signature of β-cell GA stress using existing RNA sequencing and microarray datasets generated using human islets from donors with diabetes and islets where type 1(T1D) and type 2 diabetes (T2D) had been modeled ex vivo. To narrow our results to GA-specific genes, we applied a filter set of 1,030 genes accepted as GA associated. -

Galacticomm, Inc
end of the phone line), for which dozens of For super -flexibility of incredible new multi-user games are now menu trees and ANSI screens: being developed. Upgrading from the starter system to the Entertainment Edition is quick The MenuMan Edition of The Major BBS can do everything that the starter system does, and in addition you as Sysop can create your own menu trees, with menus leading to menus lead- ing to menus, as deeply "nested" as you like. O tip Old The "leaves" of your menu trees can be ordi- 1111111 MEI nary ASCII orANSIfiles, which are simply - dumped to the user's display (with or without automatic screen breaks), or they can be any of the built-in functions of the BBS such as scanning the user's incoming E -Mail or firing up a SIG quickscan. Includes commands like GO <pagename>, FIND <topic>, USERS, and for the Sysop, the equivalent of the DOS and easy and involves no loss of data or func- commandsDIR, RENAME, COPY, DEL, tion. Prerequisite: The Major BBS Standard MKDIR,andRMDIR,as well as a set of priv- ...and that's not all! For advanced applica- Edition. ileged commands for editing and extending tions, we also offer an X.25 direct -connect Entertainment extensions $149 the menu trees, remotely, while the BBS re- software option, a protected -mode develop- Entertainment C source extensions*. $129 mains fully online. Upgrading from the starter ment toolkit, and special licensing arrange- system to the MenuMan Edition takes only ments for up to 256 simultaneous users! And If your requirements include minutes. -
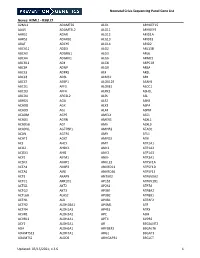
NICU Gene List Generator.Xlsx
Neonatal Crisis Sequencing Panel Gene List Genes: A2ML1 - B3GLCT A2ML1 ADAMTS9 ALG1 ARHGEF15 AAAS ADAMTSL2 ALG11 ARHGEF9 AARS1 ADAR ALG12 ARID1A AARS2 ADARB1 ALG13 ARID1B ABAT ADCY6 ALG14 ARID2 ABCA12 ADD3 ALG2 ARL13B ABCA3 ADGRG1 ALG3 ARL6 ABCA4 ADGRV1 ALG6 ARMC9 ABCB11 ADK ALG8 ARPC1B ABCB4 ADNP ALG9 ARSA ABCC6 ADPRS ALK ARSL ABCC8 ADSL ALMS1 ARX ABCC9 AEBP1 ALOX12B ASAH1 ABCD1 AFF3 ALOXE3 ASCC1 ABCD3 AFF4 ALPK3 ASH1L ABCD4 AFG3L2 ALPL ASL ABHD5 AGA ALS2 ASNS ACAD8 AGK ALX3 ASPA ACAD9 AGL ALX4 ASPM ACADM AGPS AMELX ASS1 ACADS AGRN AMER1 ASXL1 ACADSB AGT AMH ASXL3 ACADVL AGTPBP1 AMHR2 ATAD1 ACAN AGTR1 AMN ATL1 ACAT1 AGXT AMPD2 ATM ACE AHCY AMT ATP1A1 ACO2 AHDC1 ANK1 ATP1A2 ACOX1 AHI1 ANK2 ATP1A3 ACP5 AIFM1 ANKH ATP2A1 ACSF3 AIMP1 ANKLE2 ATP5F1A ACTA1 AIMP2 ANKRD11 ATP5F1D ACTA2 AIRE ANKRD26 ATP5F1E ACTB AKAP9 ANTXR2 ATP6V0A2 ACTC1 AKR1D1 AP1S2 ATP6V1B1 ACTG1 AKT2 AP2S1 ATP7A ACTG2 AKT3 AP3B1 ATP8A2 ACTL6B ALAS2 AP3B2 ATP8B1 ACTN1 ALB AP4B1 ATPAF2 ACTN2 ALDH18A1 AP4M1 ATR ACTN4 ALDH1A3 AP4S1 ATRX ACVR1 ALDH3A2 APC AUH ACVRL1 ALDH4A1 APTX AVPR2 ACY1 ALDH5A1 AR B3GALNT2 ADA ALDH6A1 ARFGEF2 B3GALT6 ADAMTS13 ALDH7A1 ARG1 B3GAT3 ADAMTS2 ALDOB ARHGAP31 B3GLCT Updated: 03/15/2021; v.3.6 1 Neonatal Crisis Sequencing Panel Gene List Genes: B4GALT1 - COL11A2 B4GALT1 C1QBP CD3G CHKB B4GALT7 C3 CD40LG CHMP1A B4GAT1 CA2 CD59 CHRNA1 B9D1 CA5A CD70 CHRNB1 B9D2 CACNA1A CD96 CHRND BAAT CACNA1C CDAN1 CHRNE BBIP1 CACNA1D CDC42 CHRNG BBS1 CACNA1E CDH1 CHST14 BBS10 CACNA1F CDH2 CHST3 BBS12 CACNA1G CDK10 CHUK BBS2 CACNA2D2 CDK13 CILK1 BBS4 CACNB2 CDK5RAP2 -

Table of Contents
Table of Contents 1. - EXAMINING ATTITUDES AND WILLINGNESS TO PAY FOR AQUACULTURED SEAFOOD ATTRIBUTES I. Ko Britwum * II. Caroline Noblet 2. - Development of a Hybrid Thermoplastic Composite and Concrete Deck System I. Benjamin Smith * II. William Davids 3. - Undergraduate Nursing Students’ Perspectives and Attitudes Caring for Elderly Patients at End of Life I. Karen Chase * II. Patricia Poirier 4. - Backpack Programs: How Maine Elementary Schools Are Tackling Childhood Hunger I. Julianna Acheson * II. Julia Van Steenberge III. Dean Rando IV. Ashlee Atchinson V. Sandra Caron 5. - Using Structure from Motion and 3D Printing as a Method for Preserving the Petroglyphs of Machias Bay, Maine I. Kendra Bird * II. Lisa Neuman 6. - Capacity Assessment of Older T-Beam Bridges Using Field Load Testing and Nonlinear Proxy Finite-Element Analysis I. Andrew Schanck * II. William Davids 7. - Interventions Supporting Social Communication Skills in Preschool-Aged Children with Autism Spectrum Disorder I. Paige Hanson * II. Paige Castonguay III. Heather Lowry IV. Taylor Dupont V. Paige Lane 8. - Attitudes Towards Immigration Following the 2018 Family Separation Crisis: Content Analysis of Tweets in The Washington Post vs Fox News I. Rebecca Blodgett * II. Vincent Eze III. Ariana Cruwys IV. Sandra Caron 9. - Nurses Role in Central Line-Associated Bloodstream Infection Prevention I. Laura Roberts * II. Julia Schnee III. Bronwyn West IV. Alex Roderick V. Valerie Herbert 10. - Overwintering strategies of the salmon louse Lepeophtheirus salmonis I. Emma Taccardi * II. Carrie Byron III. Ian Bricknell 11. - The eects of diverse aged enrollment on community school literacy rates in rural Zambia: Case study on Impact Network International schools, Eastern Province Zambia I. -

Induced Early Expression of Mrf4 but Not Myog Rescues Myogenesis in the Myod/Myf5 Double- Morphant Zebrafish Embryo
Research Article 481 Induced early expression of mrf4 but not myog rescues myogenesis in the myod/myf5 double- morphant zebrafish embryo Esther Schnapp1,*, Anna Silvia Pistocchi2,*, Evangelia Karampetsou2, Efrem Foglia2, Carla Lora Lamia2, Franco Cotelli2,‡ and Giulio Cossu1,2,‡ 1Stem Cell Research Institute, DiBiT, San Raffaele Scientific Institute, 58 via Olgettina, 20132 Milan, Italy 2Department of Biology, University of Milan, 26 via Celoria, 20133 Milan, Italy *These authors contributed equally to this work ‡Authors for correspondence (e-mails: [email protected]; [email protected]) Accepted 13 October 2008 Journal of Cell Science 122, 481-488 Published by The Company of Biologists 2009 doi:10.1242/jcs.038356 Summary Muscle regulatory factors activate myogenesis in all vertebrates, inhibition, we were able to investigate how myogenesis occurs in but their role has been studied in great detail only in the mouse the absence of a myotome. We report that in the complete absence embryo, where all but myogenin – Myod, Myf5 and Mrf4 – are of a myotome, subsequent myogenesis is abolished, whereas sufficient to activate (albeit not completely) skeletal myogenesis. myogenesis does proceed, albeit abnormally, when the In the zebrafish embryo, myod and myf5 are required for morpholino inhibition was not complete. Therefore our data also induction of myogenesis because their simultaneous ablation show that the early myotome is essential for subsequent skeletal prevents muscle development. Here we show that mrf4 but not muscle differentiation and patterning in the zebrafish. myog can fully rescue myogenesis in the myod/myf5 double morphant via a selective and robust activation of myod, in keeping Supplementary material available online at with its chromatin-remodelling function in vitro. -

Characterization of Ncf1 Mutants in a Zebrafish Model of Innate Immune Function with Human Influenza a Virus Infection
The University of Maine DigitalCommons@UMaine Honors College Spring 5-2020 Characterization of ncf1 Mutants in a Zebrafish Model of Innate Immune Function with Human Influenza A Virus Infection Lily Charpentier Follow this and additional works at: https://digitalcommons.library.umaine.edu/honors Part of the Immunology and Infectious Disease Commons, Influenza Humans Commons, and the Virus Diseases Commons This Honors Thesis is brought to you for free and open access by DigitalCommons@UMaine. It has been accepted for inclusion in Honors College by an authorized administrator of DigitalCommons@UMaine. For more information, please contact [email protected]. CHARACTERIZATION OF NCF1 MUTANTS IN A ZEBRAFISH MODEL OF INNATE IMMUNE FUNCTION WITH HUMAN INFLUENZA A VIRUS INFECTION by Lily Charpentier A Thesis Submitted to Partial Fulfillment of the Requirements for a Degree with Honors (Biochemistry) The Honors College The University of Maine May 2020 Advisory Committee: Benjamin L. King, Assistant Professor of Bioinformatics, Advisor Edward Bernard, Lecturer & Undergraduate Coordinator of Molecular & Biomedical Sciences R.W. Estela, Honors Preceptor Sally D. Molloy, Assistant Professor of Genomics Robert Wheeler, Associate Professor of Microbiology ABSTRACT Seasonal influenza A virus (IAV) infections and their associated respiratory diseases are the cause of an estimated 650,000 deaths each year, according to the World Health Organization. The zebrafish (Danio rerio) is a powerful vertebrate model to study innate immune function and host-pathogen interactions as the function of neutrophils and other phagocytes can be characterized in vivo. Preliminary studies have shown an increase in neutrophil respiratory burst activity to eliminate the invading pathogen, yet little is known of all of the mechanisms involved in neutrophil function. -

CENTOGENE's Curated Gene List of Pathogenic and Likely Pathogenic
CENTOGENE’s curated gene list of pathogenic and likely pathogenic variants indicated in severe and early-onset disorders VALID FROM 01.08.2020 AAAS, AARS1, AARS2, ABAT, ABCA12, ABCA3, ABCB11, ABCB4, ABCB7, ABCC6, ABCC8, ABCC9, ABCD1, ABCD4, ABHD12, ABHD5, ACACA, ACAD9, ACADM, ACADS, ACADVL, ACAN, ACAT1, ACE, ACO2, ACOX1, ACP5, ACSL4, ACTA1, ACTA2, ACTB, ACTG1, ACTL6B, ACVR2B, ACVRL1, ACY1, ADA, ADAM17, ADAMTS2, ADAMTSL2, ADAR, ADARB1, ADAT3, ADCY5, ADGRG1, ADGRG6, ADGRV1, ADK, ADNP, ADPRHL2, ADSL, AFF2, AFG3L2, AGA, AGK, AGL, AGPAT2, AGPS, AGRN, AGT, AGTPBP1, AGTR1, AGXT, AHCY, AHDC1, AHI1, AIFM1, AIMP1, AIPL1, AIRE, AK2, AKR1D1, AKT2, AKT3, ALAD, ALDH18A1, ALDH1A3, ALDH3A2, ALDH4A1, ALDH5A1, ALDH6A1, ALDH7A1, ALDOA, ALDOB, ALG1, ALG11, ALG12, ALG13, ALG14, ALG2, ALG3, ALG6, ALG8, ALG9, ALOX12B, ALPL, ALX3, ALX4, AMACR, AMER1, AMN, AMPD1, AMPD2, AMT, ANK2, ANK3, ANKH, ANKRD11, ANKS6, ANO10, ANO5, ANOS1, ANTXR1, ANTXR2, AP1B1, AP1S1, AP1S2, AP3B1, AP3B2, AP4B1, AP4E1, AP4M1, AP4S1, APC2, APTX, AR, ARCN1, ARFGEF2, ARG1, ARHGAP31, ARHGDIA, ARHGEF9, ARID1A, ARID1B, ARID2, ARL13B, ARL3, ARL6, ARL6IP1, ARMC4, ARMC9, ARSA, ARSB, ARSL, ARV1, ARX, ASAH1, ASCC1, ASH1L, ASL, ASNS, ASPA, ASPH, ASPM, ASS1, ASXL1, ASXL2, ASXL3, ATAD3A, ATCAY, ATIC, ATL1, ATOH7, ATP13A2, ATP1A1, ATP1A2, ATP1A3, ATP2B3, ATP5MD, ATP6AP2, ATP6V0A2, ATP6V0A4, ATP6V1A, ATP6V1B1, ATP6V1B2, ATP7A, ATP7B, ATP8A2, ATP8B1, ATRX, AUH, AUTS2, B3GALNT2, B3GALT6, B3GAT3, B3GLCT, B4GALNT1, B4GALT7, B4GAT1, B9D1, B9D2, BANF1, BBS1, BBS10, BBS12, BBS2, BBS4, BBS5, BBS7, BBS9, BCAP31, -

C12) United States Patent (10) Patent No.: US 7,272,639 Bl Levergood Et Al
111111 1111111111111111111111111111111111111111111111111111111111111 US007272639Bl c12) United States Patent (10) Patent No.: US 7,272,639 Bl Levergood et al. (45) Date of Patent: *Sep.18,2007 (54) INTERNET SERVER ACCESS CONTROL 4,578,530 A 3/1986 Zeidler .................... 178/22.09 AND MONITORING SYSTEMS 4,734,858 A 3/1988 Schlafly 4,755,940 A 7/1988 Bracht! et a!. (75) Inventors: Thomas Mark Levergood, Hopkinton, 4,759,063 A 7/1988 Chaum MA (US); Lawrence C. Stewart, 4,759,064 A 7/1988 Chaum 4,775,935 A 10/1988 Yourick Burlington, MA (US); Stephen Jeffrey 4,795,890 A 111989 Goldman Morris, Westford, MA (US); Andrew 4,799,156 A 111989 Shavit eta!. C. Payne, Lincoln, MA (US); George 4,812,628 A 3/1989 Boston eta!. Winfield Treese, Newton, MA (US) 4,827,508 A 5/1989 Shear 4,891,503 A 111990 Jewell (73) Assignee: Soverain Software LLC, Chicago, IL (US) (Continued) FOREIGN PATENT DOCUMENTS ( *) Notice: Subject to any disclaimer, the term of this patent is extended or adjusted under 35 EP 0172670 2/1986 U.S.C. 154(b) by 0 days. (Continued) This patent is subject to a terminal dis OTHER PUBLICATIONS claimer. T. Berners-Lee et al., RFC 1738: Uniform Resource Locators (21) Appl. No.: 09/005,479 (URLs), Network Working Group, Dec. 1994, pp. 1-25.* (Continued) (22) Filed: Jan. 12, 1998 Primary Examiner-Patrice Winder Related U.S. Application Data (74) Attorney, Agent, or Firm-Jones Day (63) Continuation of application No. 08/474,096, filed on (57) ABSTRACT Jun. 7, 1995, now Pat. -

The Ciliopathy Gene Nphp-2 Functions in Multiple Gene Networks and Regulates
THE CILIOPATHY GENE NPHP-2 FUNCTIONS IN MULTIPLE GENE NETWORKS AND REGULATES CILIOGENESIS IN C. ELEGANS By SIMON ROBERT FREDERICK WARBURTON-PITT A dissertation submitted to the Graduate School – New Brunswick and The Graduate School of Biomedical Sciences Rutgers, The State University of New Jersey In partial fulfillment of the requirements For the degree of Doctor of Philosophy Graduate Program in Molecular Genetics and Microbiology Written under the direction of Maureen M. Barr, PhD And approved by New Brunswick, New Jersey January, 2015 © 2015 Simon Warburton-Pitt ALL RIGHTS RESERVED ABSTRACT OF THE DISSERTATION The ciliopathy gene nphp-2 functions in multiple gene networks and regulates ciliogenesis in C. elegans By SIMON ROBERT FREDERICK WARBURTON-PITT Dissertation Director: Maureen Barr Cilia are hair-like organelles that function as cellular antennae. Cilia are conserved across eukaryotes, and play a vital role in many biological processes including signal transduction, signal cascades, cell-cell signaling, cell orientation, cell-cell adhesion, motility, interorganismal communication, building extracellular matrix, and inducing fluid flow. In humans, cilia are present in a majority of tissue types, and cilia dysfunction can lead to a range of syndromic ciliopathies, including nephronopthisis (NPHP) and Meckel Syndrome (MKS). Cilia have a microtubule backbone, the axoneme, and are composed of multiple subcompartments, each with a specific function and composition: the transition zone (TZ) anchoring to the axoneme to the membrane, the doublet region extending from the TZ, and in some cilia types, the singlet region extending from the doublet region. The nematode C. elegans is a well-established model of cilia biology, and possesses cilia at the distal end of sensory dendrites.